دوره 33، شماره 6 - ( شهریور 1404 )
جلد 33 شماره 6 صفحات 9134-9124 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Kalhor H, Siroosi M. Identification of Phenazines with Potential Inhibitory Activity against OXA-48 Carbapenemase by Using Molecular Docking Approach to Combat Antibiotic Resistance. JSSU 2025; 33 (6) :9124-9134
URL: http://jssu.ssu.ac.ir/article-1-6375-fa.html
URL: http://jssu.ssu.ac.ir/article-1-6375-fa.html
کلهر حوریه، سیروسی مریم. شناسایی فنازینهای با عملکرد بالقوه مهارکنندگی آنزیم کارباپنماز OXA-48 با رویکرد داکینگ مولکولی جهت مقابله با مقاومت آنتیبیوتیکی. مجله علمي پژوهشي دانشگاه علوم پزشكي شهید صدوقی يزد. 1404; 33 (6) :9124-9134
متن کامل [PDF 1033 kb]
(168 دریافت)
| چکیده (HTML) (446 مشاهده)
متن کامل: (117 مشاهده)
مقدمه
آنتیبیوتیکهای کارباپنم خط آخر درمانی برای عفونتهای مقاوم به چند دارو هستند و به همین دلیل سازمان جهانی بهداشت در سال 2019 کارباپنمها را به عنوان آنتیبیوتیکهای بسیار مهم طبقهبندی کرد (1). انتروباکتریاسهها از جمله باکتریهای مهمی هستند که مقاومت به کارباپنمها در آنها چالشهای درمانی قابلتوجهی ایجاد کرده است. این باکتریها با تولید کارباپنماز، کارباپنمها را هیدرولیز میکنند (2). کارباپنمازها شامل بتالاکتامازهای کلاسهای A و D که دارای ریشه آمینواسیدی سرین در جایگاه فعال خود هستند (1) و تمام متالوبتالاکتامازهای کلاس B هستند (3). در خاورمیانه، Klebsiella pneumoniae شایعترین انتروباکتریاسه تولیدکننده کارباپنماز است و در میان کارباپنمازها، کارباپنماز OXA-48شایعترین کارباپنماز یافت شده در خاورمیانه و ایران است (4). یک راهکار برای درمان عفونتهای انتروباکتریاسههای مقاوم به کارباپنم، ترکیب یک بتالاکتام و یک مهارکننده بتالاکتاماز، مانند سفتازیدیم-آویباکتام است، اگرچه ایجاد مقاومت استفاده از آن را محدود کرده است. مروپنم-وابرباکتام ترکیب درمانی دیگری است که علیه کارباپنمازهای کلاس B و D که اتفاقاً در ایران و سایر کشورهای خاورمیانه شایعتر از کلاس A هستند، فعال نیست (5). این واقعیتها نشاندهنده نیاز به یافتن یک مهارکننده است که با غیرفعال کردن کارباپنماز OXA-48 بتواند فعالیت کارباپنمها، این آنتیبیوتیکهای بسیار مهم، را بازگرداند (1). در حال حاضر، استفاده از متابولیتهای ثانویه باکتریایی برای اهداف ضد میکروبی بهطور فزایندهای مورد توجه قرار گرفته است. از جمله متابولیتهای میکروبی میتوان به فنازینها اشاره کرد. ترکیبات فنازین دستهای از ترکیبات طبیعی هستند که بهعنوان متابولیتهای ثانویه توسط برخی باکتریها، به ویژه گونههای Pseudomonas و Streptomyces تولید میشوند (6). فنازینها به دلیل ساختار شیمیایی خاص خود، توانایی تعامل با DNA و مهار آنزیمهای کلیدی مانند توپوایزومرازها را دارند. فنازینها دارای یک هسته آروماتیک دو حلقهای هستند که میتواند با گروههای مختلفی مانند هیدروکسیل، کربوکسیل، متوکسی و آمینو جایگزین شود. برخی از فنازینها دارای زنجیرههای جانبی پیچیده مانند ترپنوئیدها یا کربوهیدراتها هستند که به آنها خواص بیولوژیکی خاصی میبخشد. ترکیبات فنازین به دلیل خواص بیولوژیکی متنوع و پتانسیل بالای آنها در درمان بیماریها، موضوع تحقیقات گسترده در شیمی دارویی و بیوتکنولوژی هستند. این ترکیبات دارای خواص بیولوژیکی متنوعی از جمله فعالیتهای ضد میکروبی، ضد توموری، ضد مالاریا و ضد انگلی هستند (7). تاکنون ترکیبات مشتق شده از فنازینها مانند کلورافین و پیوسیانین به دلیل فعالیت ضد میکروبی قوی خود مورد مطالعه قرار گرفتهاند. همچنین برخی از مشتقات سنتزی فنازینها به عنوان مهارکنندههای توپوایزومراز در درمان سرطان بررسی شدهاند. ترکیباتی مانند DACA (Acridine-4-carboxamide) و مشتقات فنازینی آن به عنوان عوامل ضد توموری قوی شناخته شدهاند. بهعلاوه کلوفازیمین (Clofazimine)به عنوان یک ترکیب سنتزی فنازین در درمان بیماریهایی مانند جذام و سل مورد استفاده قرار گرفته است. کلوفازیمین با تحریک آزادسازی رادیکالهای آزاد توسط گلبولهای سفید، اثرات ضد میکروبی خود را اعمال میکند (7،8). یکی از راه-های شناسایی ترکیبات مهار کننده آنزیمهای کارباپنماز، رویکرد داکینگ مولکولی است. داکینگ مولکولی به عنوان یک روش محاسباتی برای پیشبینی برهمکنشهای لیگاند-پروتئین، از دهه ۱۹۸۰ توسعه یافته است. این روش به تدریج با پیشرفتهای محاسباتی و الگوریتمی، به یکی از ابزارهای اصلی در طراحی دارو و کشف دارو تبدیل شده است (9). با استفاده از این رویکرد میتوان تعداد زیادی از ترکیبات موجود در پایگاههای داده¬ را علیه هدف اختصاصی بهصورت مجازی غربالگری کرد. این روش در زمان و هزینه صرفهجویی میکند و سبب معرفی تعداد زیادی از ترکیباتی که بالقوه علیه هدف اختصاصی مورد نظر موثر هستند میشود (10). در همین راستا، مطالعهی حاضر بر شناسایی فنازینهای با عملکرد مهاری کارباپنماز OXA-48 با رویکرد غربالگری مجازی تمرکز دارد. در این پژوهش با استفاده از داکینگ مولکولی برهمکش بین آنزیم کارباپنمازOXA-48 و ترکیبات فنازینی بررسی شد. در ادامه، نتایج انرژی اتصال و تعامل مولکولی بین آنزیم کارباپنماز OXA-48 و ترکیبات فنازین بررسی شدند، تا ترکیبی با توانایی مهاری بالقوه علیه آنزیم کارباپنماز OXA-48 شناسایی و به عنوان نامزد دارویی موثر برای بهرهبرداری بالینی معرفی گردد.
روش بررسی
انتخاب و آمادهسازی پروتئین: ساختار کریستالی آنزیم کارباپنماز OXA-48 با کد شناسایی PDB: 3HBR از بانک اطلاعات پروتئین (PDB) دریافت شد (11). در ادامه، نرمافزار AutoDock Tools4.2 برای آمادهسازی آنزیم کارباپنماز OXA-48 به عنوان گیرنده جهت مطالعات داکینگ مولکولی مورد استفاده قرار گرفت (12). به این ترتیب که مولکولهای آب، کوفاکتورها و سایر مولکولهایی که در جایگاه فعال گیرنده حضور ندارند، حذف شدند. اتمها با انواع اتمهای AutoDock تنظیم شده و سپس ترتیب پیوندها، اتمهای هیدروژن و بارهای Gasteiger-Marsili به ساختار پروتئین اضافه شدند. در نهایت، ساختار گیرنده در قالب PDBQT آماده شد (12). انتخاب و آمادهسازی جایگاههای اتصال آنزیم کارباپنمازOXA-48 برای انجام مطالعات داکینگ مولکولی به منظور تعیین ریشههای آمینواسیدی در جایگاه فعال آنزیم کارباپنماز OXA-48 که در انجام واکنش آنزیمی نقش دارند، فایل3HBR.pbb با استفاده از سرور PDBsum و نرمافزار LigPlot+ مورد مطالعه قرار گرفت (13). بر این اساس، ریشههای آمینواسیدی Ser118، Thr209، Arg214، Arg250، Ser70، KCX 73، Gln 124، Ile 102 و Ser244به عنوان جایگاههای اتصال برای برای اتصال انعطافپذیر انتخاب شدند. سپس نرمافزار AutoDock Tools4.2 برای پیش بینی جعبه شبکه در نقاط شبکه X = 26 Å، Y = 26 Å و Z = 26 Å استفاده شد و فاصله شبکه Å1 در نظر گرفته شد.
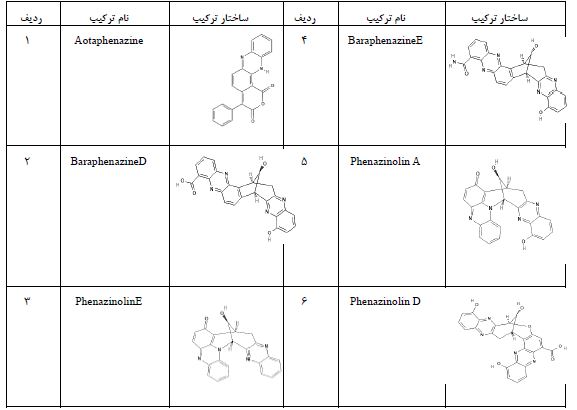
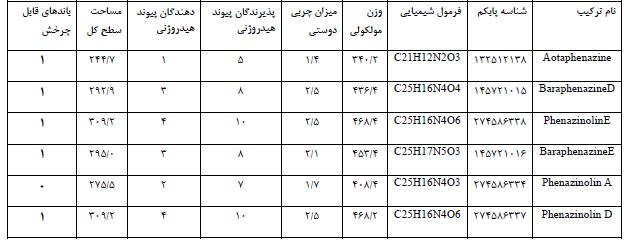
انتخاب و آماده سازی لیگاند: جهت انتخاب لیگاندها مقالات علمی مرتبط با متابولیتهای ثانویه فنازینها بررسی گردید. بر اساس متون معتبر علمی، متابولیتهای ثانویه فنازینها به دلیل ساختار شیمیایی منحصربهفرد دارای فعالیت بیولوژیکی و پتانسیل بالای اتصال به پروتئینهای هدف هستند و به دلیل ویژگیهای ضد میکروبی اثبات شدهشان، به عنوان لیگاندهای مناسب برای این مطالعه انتخاب شدند. اگرچه برخی از ترکیبات فنازینی پیشتر فعالیتهای زیستی از جمله اثرات ضد میکروبی نشان داده اند (14-17، 8)، اما هدف از انجام داکینگ مولکولی در این مطالعه، بررسی دقیقتر مکانیسم اتصال این ترکیبات به آنزیم کارباپنماز OXA-48 و درک مکانیسم عمل احتمالی این ترکیبات در سطح مولکولی و همچنین شناسایی ترکیباتی با پتانسیل مهارکنندگی بالاتر میباشد. بنابراین بر اساس مطالعات انجام شده، شش مولکول کوچک به نامهای Aotaphenazine، BaraphenazineD، PhenazinolinE، BaraphenazineE، PhenazinolinA و PhenazinolinD متعلق به خانواده فنازینها به عنوان لیگاند برای این مطالعه انتخاب شدند. ساختار دو بعدی این لیگاندها از پایگاه داده Pubchem (https://pubchem.ncbi.nlm.nih.gov/) با پسوند sdf دریافت شد و با استفاده از نرمافزارOpenBabel 2.3.2 به پسوند pdb تغییر پیدا کرد (18). بهینه سازی لیگاندها توسط نرمافزار Chem Draw(3D) (نسخه 17.1) انجام شد (https://perkinelmer-chemdraw-professional.software.informer.com/17.1). در نهایت ترکیبات بهینهسازی شده جهت انجام داکینگ مولکولی توسط نرمافزار Autodock tools به پسوند pdbqt تغییر پیدا کردند. نام و ساختار این ترکیبات در جدول 1 نشان داده شده است.
مطالعه داکینگ مولکولی: به منظور انجام داکینگ مولکولی از نرم افزار AutoDock Vina استفاده شد (19). به این صورت که داکینگ برای جایگاه اتصال تعریف شده آنزیم کارباپنماز OXA-48 به عنوان گیرنده و لیگاندهای آماده شده بهطور جداگانه انجام شد. انرژی تمایل لیگاندها به گیرنده به صورت کیلوکالری بر مول محاسبه شد، بهطوری که هرچه مقدار انرژی کمتر باشد تمایل لیگاند به گیرنده بیشتر است. نتایج اتصال و تعامل مولکولی بین آنزیم کارباپنماز OXA-48 و لیگاندها با استفاده از نرمافزار Pymol و LigPlot+ ارزیابی و مشاهده شد (13،20).
نتایج
تجزیه و تحلیل ساختار کارباپنماز OXA-48: ارزیابی ساختار آنزیم کارباپنماز OXA-48 نشان داد که این آنزیم یک هومودایمر با چهار زنجیره است که هر زنجیره 256 ریشهی آمینواسیدی دارد. جایگاه فعال این آنزیم در یک کانال باریک با عرض، عمق و طول 5، 10 و Å20 قرار گرفته است. این کانال توسط چهار ریشهی آمینواسیدی Arg214، Gln124، Ile102 و Ser244 احاطه شده است. بر اساس مقایسه با سایر بتالاکتامازهای کلاس D، ریشههای آمینواسیدی Ser70، Lys73 (لیزین کاربامیله شده) و Arg250 که در جایگاه فعال آنزیم قرار دارند، حفاظت شده هستند (شکل 1).
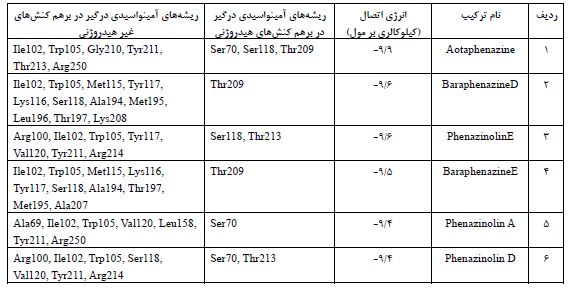
ارزیابی برهمکنش آنزیم کارباپنماز OXA-48 با فنازینها: اتصال مولکولی بین آنزیم کارباپنماز OXA-48 و فنازینها توسط Autodock Vina انجام شد. نتایج اتصال مولکولی نشان داد که تمام لیگاندها میل اتصال منفی به پروتئین هدف داشتند (جدول 2). محدودهی انرژی تمایل لیگاندها به گیرنده از 9/9- تا 4/9- کیلوکالری بر مول بود، به این صورت که بالاترین و کمترین انرژی تمایل به ترتیب مربوط به ترکیب Aotaphenazine و Phenazinolin D تعیین شد. ریشههای آمینواسیدی درگیر در پیوندهای هیدروژنی و غیر هیدروژنی با هرکدام از لیگاندها که از طریق بررسی نتایج داکینگ مولکولی توسط نرم افزار LigPlot+ تعیین شده با جزئیات بیشتر در جدول 2 و شکل 2 آورده شده است.
دارا بودن خصوصیات فیزیکوشیمیایی مطلوب در کنار اثربخشی دارویی از جمله شاخصهای مهم جهت ارزیابی یک مولکول به عنوان یک نامزد دارویی مناسب است. لذا در این مطالعه خصوصیات فیزیکوشیمیایی ترکیبات فنازینی انتخاب شده از طریق پایگاه داده PubChem (https://pubchem.ncbi.nlm.nih.gov/) بررسی گردید.
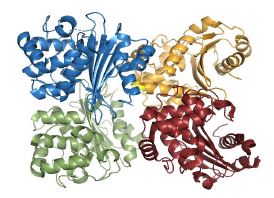
شکل 1: ساختار سه بعدی آنزیم کارباپنماز OXA-48 (با کد شناسایی: PDB: 3HBR)
جدول:1 نام و ساختار ترکیبات فنازین مورد استفاده در این مطالعه
جدول 2: ارزیابی انرژی اتصال (کیلوکالری بر مول) ترکیبات فنازین به آنزیم کارباپنماز OXA-48 و بررسی ریشههای آمینواسیدی درگیر در پیوندهای هیدروژنی و غیرهیدروژنی.
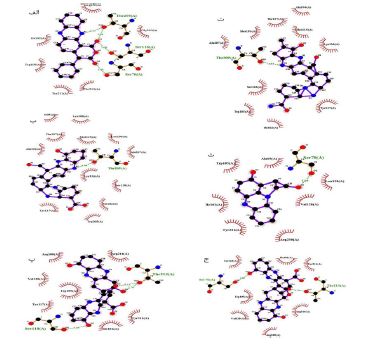
شکل 2: ساختار دو بعدی از برهمکنش آمینواسیدهای آنزیم کارباپنماز OXA-48 درگیر در پیوندهای هیدروژنی و غیرهیدروژنی با ترکیبات فنازینی. آمینواسیدهای درگیر در پیوندهای هیدروژنی با رنگ سیاه، آمینواسیدهای درگیر در پیوندهای غیر هیدروژنی با رنگ قرمز و طول پیوندهای هیدروژنی به صورت خط چین سبز رنگ نشان داده شده است. الف: Aotaphenazine، ب: BaraphenazineD، پ: PhenazinolinE، ت: BaraphenazineE، ث: Phenazinolin A، ج: Phenazinolin D
جدول 3: ارزیابی خواص فیزیکوشیمیایی ترکیبات فنارینها بر اساس نرمافزار OSIRIS Data Warrior
بحث
انتروباکتریاسههای مقاوم به کارباپنم اکنون به یک معضل جهانی در زمینه درمان عفونتهای این خانواده تبدیل شدهاند، زیرا دیگر به کارباپنمها که آخرین خط درمانی آنتیبیوتیکها در بسیاری از بیمارستانها هستند، حساسیت نشان نمیدهند. در حالیکه سفتازیدیم-آویباکتام نتایج امیدوارکنندهای در درمان این عفونتها نشان داده است، اما در بسیاری از کشورها به طور گسترده استفاده نمیشود و مقاومت در برابر این ترکیب نیز در حال افزایش است. با توجه به فوریت این وضعیت، سازمان بهداشت جهانی در به روز رسانی سال 2024 خود، این باکتریها را به عنوان پاتوژنهایی که نیازمند درمانهای جدید هستند، فهرست کرده است. درک مکانیسمهای مقاومت به کارباپنمها در انتروباکتریاسهها، مانند افزایش بیان پمپهای افلاکس، جهش در پورینها و تولید کارباپنمازها، برای یافتن داروهای جدید برای درمان آنها ضروری است. در میان کارباپنمازهای تولید شده توسط انتروباکتریاسهها، OXA-48 شایعترین کارباپنماز در منطقه خاورمیانه، از جمله ایران است. با اینحال، بهترین درمان برای عفونتهای ناشی از باکتریهای تولیدکننده کارباپنماز OXA-48 هنوز مشخص نیست. سفتازیدیم- آویباکتام یک درمان تایید شده برای انتروباکتریاسههای مقاوم به کارباپنم است. با اینحال، ظهور مقاومت در برابر این درمان نگران کننده است. وابربکتام یک مهارکننده بتالاکتاماز است که در ترکیب با مروپنم برای درمان عفونتهای ناشی از انتروباکتریاسههای مقاوم به کارباپنم استفاده می شود، اما وابربکتام در برابر کارباپنماز OXA-48 عملکردی ندارد (5). برای رسیدگی به این مسئله حیاتی، در این مطالعه از رویکرد غربالگری مجازی برای شناسایی مولکولهای کوچک از خانوادهی فنازینها با توانایی مهار کارباپنماز OXA-48 استفاده شد. فنازینها، متابولیتهای ثانویهای هستند که توسط باکتریهای مختلف از جمله استرپتومایسسها تولید میشوند و خواص ضدتوموری، ضد انگلی و ضدمالاریایی آنها تأیید شده است (8). بنایراین در این مطالعه به منظور یافتن مهارکنندههای جدید کارباپنماز OXA-48، ترکیبات فنازین (متابولیتهای ثانویه گونه های استرپتومایسس) انتخاب شده و سپس با روشهای داکینگ مولکولی نحوه اتصال و برهم کنشهای این آنزیم و ترکیبات بررسی گردید. نتیجه مطالعه حاضر نشان داد که ترکیبات فنازین مورد مطالعه، میتوانند با اتصال به جایگاه فعال آنزیم کارباپنماز OXA-48، موجب مهار این آنزیم شوند. بر اساس نتایج داکینگ، پتانسیل مهاری ترکیبات مورد بررسی با یکدیگر تفاوت داشته و قویترین اتصالات با بیشترین پتانسیل مهاری مربوط به ترکیباتAotaphenazine ، PhenazinolinE و BaraphenazineD است. ترکیب Aotaphenazine با انرژی اتصال 9/9- کیلوکالری بر مول و ازطریق سه پیوند هیدروژنی با آمینواسیدهای Ser70، Ser118 و Thr209و همچنین شش پیوند هیدروفوبی با آمینواسیدهایIle102 ،Trp105 ،Gly210 ،Tyr211 ، Thr213 و Arg250برهمکنش دارد. ترکیب PhenazinolinE با انرژی اتصال 9/6- کیلوکالری بر مول ازطریق دو پیوند هیدروژنی با آمینواسیدهای Ser118 و Thr213و همچنین هفت پیوند هیدروفوبی با آمینواسیدهای Trp105، Ile102، Arg100، Tyr117،Val120 ، Tyr211 و Arg214 برهمکنش دارد. در حالیکه ترکیب BaraphenazineD با انرژی اتصال 9/6- کیلوکالری بر مول ازطریق یک پیوند هیدروژنی با آمینواسید Thr209 و یازده پیوند هیدروفوبی با آمینواسیدهایIle102 ،Trp105 ،Met115 ،Tyr117 ،Lys116 ،Ser118 ، Met195،Leu196 ،Thr197 ، Lys208 و Ala194 برهمکنش دارد (جدول 1 و شکل 2). همچنین ترکیبات BaraphenazineE ، Phenazinolin A و Phenazinolin D انرژهای اتصال مناسبی را نشان دادند که به ترتیب شامل 9/5-، 9/4- و 9/4- کیلوکالری بر مول میباشند (جدول 1). در همه موارد، ترکیبات فنازینی توسط پیوندهای هیدروژنی با حداقل یکی از این آمینواسیدهای کلیدی Ser70، Ser118 و Thr209 اتصال و برهمکنش دارند. با این حال، Aotaphenazine، PhenazinolinE و Phenazinolin D پیوندهای هیدروژنی بیشتری نسبت به سایر ترکیبات انتخاب شده نشان دادند. علیرغم تعداد پیوندهای هیدروژنی، تعاملات هیدروفوبیک بیشترین تعاملات در کمپلکسها بودند. علاوه بر این، نتایج این مطالعه نشان داد که همه ترکیبات فنازینی انتخاب شده میتوانند به طور مؤثر با آمینواسیدهای جایگاه فعال کارباپنماز OXA-48 تعامل داشته باشند و در نتیجه میتوانند از طریق مسدود کردن جایگاه اتصال کارباپنماز OXA-48، در درمان عفونت باکتریایی به همراه آنتی بیوتیکها نقش داشته باشند. اثبات شده است که حدود 90٪ از ترکیبات فعال خوراکی از قانون لیپنسکی (RO5) پیروی میکنند. قانون لیپینسکی پیشبینی میکند که ترکیباتی که وزن مولکولی کمتر از 500 دالتون، فاکتور چربی دوستی کمتر از 5، تعداد اتمهای پذیرنده هیدروژن کمتر از 10 و تعداد اتمهای دهنده هیدروژن کمتر از 5 داشته باشند دارای ویژگیهای خوراکی بودن هستند (21). لذا برای بررسی ترکیبات از لحاظ این قانون در این مطالعه، خواص فیزیکوشیمیایی مهم ترکیبات انتخاب شده توسط نرمافزار OSIRIS Data Warrior بررسی شد (22) (جدول3). نتایج نشان داد که ترکیبات انتخاب شده از قانون لیپنسکی پیروی میکنند و قابلیت خوراکی بودن را دارند. با توجه به نیاز مقابله با گسترش جهانی مقاومت آنتیبیوتیکی، در این مطالعه ترکیبات اولیه معقولی برای بررسیهای تجربی در مهار آنزیم کارباپنماز OXA-48 شناسایی گردید که ممکن است برای محققانی که به شناسایی درمانهای جدید با متابولیتهای ثانویه علاقه مند هستند، قابلتوجه باشند.
نتیجهگیری
نتایج این مطالعه نشان میدهد که ترکیبات فنازینی دارای انرژی تمایل بالا و برهمکنشهای قابل توجهی با آنزیم کارباپنماز OXA-48 بوده و همچنین خصوصیات فیزیکوشیمیایی مناسب را از خود نشان میدهند. لذا این ترکیبات که به صورت طبیعی در باکتریهای جنس Streptomyces وجود دارند، میتوانند پتانسیل بالایی برای مهار آنزیم کارباپنماز OXA-48 و مقابله با مقاومت آنتی بیوتیکی داشته باشند. پیشنهاد میشود که اثر مهاری این ترکیبات در شرایط آزمایشگاهی و بالینی نیز بررسی شود تا اطمینان بیشتری از خاصیت ضدباکتریایی آنها به دست آید.
سپاسگزاری
این مطالعه برگرفته از طرح تحقیقاتی میباشد که در معاونت تحقیقات و فناوری دانشگاه علوم پزشکی تهران و معاونت تحقیقات و فناوری دانشگاه علوم پزشکی قم به ترتیب با کد 71572-101-1-1403 و 14031849 به ثبت رسیده است. بدین وسیله نویسندگان مقاله مراتب قدردانی و سپاس خود را از تمام افرادی که در این مطالعه همکاری داشته اند اعلام مینمایند.
حامی مالی: حامیان مالی این مطالعه دانشگاه علوم پزشکی تهران و دانشگاه علوم پزشکی قم
تعارض در منافع: وجود ندارد.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه علوم پزشکی تهران و دانشگاه علوم پزشکی قم تایید شده است (کد 1403.100. IR.TUMS.MEDICINE.REC.).
مشارکت نویسندگان
حوریه کلهر و مریم سیروسی در ارائه ایده حوریه کلهر و مریم سیروسی در طراحی مطالعه، حوریه کلهر و مریم سیروسی در جمعآوری دادهها، حوریه کلهر و مریم سیروسی در تجزیه و تحلیل دادهها مشارکت داشته و همه نویسندگان در تدوین، ویرایش اولیه و نهایی مقاله و پاسخگویی به سوالات مرتبط با مقاله سهیم هستند.
آنتیبیوتیکهای کارباپنم خط آخر درمانی برای عفونتهای مقاوم به چند دارو هستند و به همین دلیل سازمان جهانی بهداشت در سال 2019 کارباپنمها را به عنوان آنتیبیوتیکهای بسیار مهم طبقهبندی کرد (1). انتروباکتریاسهها از جمله باکتریهای مهمی هستند که مقاومت به کارباپنمها در آنها چالشهای درمانی قابلتوجهی ایجاد کرده است. این باکتریها با تولید کارباپنماز، کارباپنمها را هیدرولیز میکنند (2). کارباپنمازها شامل بتالاکتامازهای کلاسهای A و D که دارای ریشه آمینواسیدی سرین در جایگاه فعال خود هستند (1) و تمام متالوبتالاکتامازهای کلاس B هستند (3). در خاورمیانه، Klebsiella pneumoniae شایعترین انتروباکتریاسه تولیدکننده کارباپنماز است و در میان کارباپنمازها، کارباپنماز OXA-48شایعترین کارباپنماز یافت شده در خاورمیانه و ایران است (4). یک راهکار برای درمان عفونتهای انتروباکتریاسههای مقاوم به کارباپنم، ترکیب یک بتالاکتام و یک مهارکننده بتالاکتاماز، مانند سفتازیدیم-آویباکتام است، اگرچه ایجاد مقاومت استفاده از آن را محدود کرده است. مروپنم-وابرباکتام ترکیب درمانی دیگری است که علیه کارباپنمازهای کلاس B و D که اتفاقاً در ایران و سایر کشورهای خاورمیانه شایعتر از کلاس A هستند، فعال نیست (5). این واقعیتها نشاندهنده نیاز به یافتن یک مهارکننده است که با غیرفعال کردن کارباپنماز OXA-48 بتواند فعالیت کارباپنمها، این آنتیبیوتیکهای بسیار مهم، را بازگرداند (1). در حال حاضر، استفاده از متابولیتهای ثانویه باکتریایی برای اهداف ضد میکروبی بهطور فزایندهای مورد توجه قرار گرفته است. از جمله متابولیتهای میکروبی میتوان به فنازینها اشاره کرد. ترکیبات فنازین دستهای از ترکیبات طبیعی هستند که بهعنوان متابولیتهای ثانویه توسط برخی باکتریها، به ویژه گونههای Pseudomonas و Streptomyces تولید میشوند (6). فنازینها به دلیل ساختار شیمیایی خاص خود، توانایی تعامل با DNA و مهار آنزیمهای کلیدی مانند توپوایزومرازها را دارند. فنازینها دارای یک هسته آروماتیک دو حلقهای هستند که میتواند با گروههای مختلفی مانند هیدروکسیل، کربوکسیل، متوکسی و آمینو جایگزین شود. برخی از فنازینها دارای زنجیرههای جانبی پیچیده مانند ترپنوئیدها یا کربوهیدراتها هستند که به آنها خواص بیولوژیکی خاصی میبخشد. ترکیبات فنازین به دلیل خواص بیولوژیکی متنوع و پتانسیل بالای آنها در درمان بیماریها، موضوع تحقیقات گسترده در شیمی دارویی و بیوتکنولوژی هستند. این ترکیبات دارای خواص بیولوژیکی متنوعی از جمله فعالیتهای ضد میکروبی، ضد توموری، ضد مالاریا و ضد انگلی هستند (7). تاکنون ترکیبات مشتق شده از فنازینها مانند کلورافین و پیوسیانین به دلیل فعالیت ضد میکروبی قوی خود مورد مطالعه قرار گرفتهاند. همچنین برخی از مشتقات سنتزی فنازینها به عنوان مهارکنندههای توپوایزومراز در درمان سرطان بررسی شدهاند. ترکیباتی مانند DACA (Acridine-4-carboxamide) و مشتقات فنازینی آن به عنوان عوامل ضد توموری قوی شناخته شدهاند. بهعلاوه کلوفازیمین (Clofazimine)به عنوان یک ترکیب سنتزی فنازین در درمان بیماریهایی مانند جذام و سل مورد استفاده قرار گرفته است. کلوفازیمین با تحریک آزادسازی رادیکالهای آزاد توسط گلبولهای سفید، اثرات ضد میکروبی خود را اعمال میکند (7،8). یکی از راه-های شناسایی ترکیبات مهار کننده آنزیمهای کارباپنماز، رویکرد داکینگ مولکولی است. داکینگ مولکولی به عنوان یک روش محاسباتی برای پیشبینی برهمکنشهای لیگاند-پروتئین، از دهه ۱۹۸۰ توسعه یافته است. این روش به تدریج با پیشرفتهای محاسباتی و الگوریتمی، به یکی از ابزارهای اصلی در طراحی دارو و کشف دارو تبدیل شده است (9). با استفاده از این رویکرد میتوان تعداد زیادی از ترکیبات موجود در پایگاههای داده¬ را علیه هدف اختصاصی بهصورت مجازی غربالگری کرد. این روش در زمان و هزینه صرفهجویی میکند و سبب معرفی تعداد زیادی از ترکیباتی که بالقوه علیه هدف اختصاصی مورد نظر موثر هستند میشود (10). در همین راستا، مطالعهی حاضر بر شناسایی فنازینهای با عملکرد مهاری کارباپنماز OXA-48 با رویکرد غربالگری مجازی تمرکز دارد. در این پژوهش با استفاده از داکینگ مولکولی برهمکش بین آنزیم کارباپنمازOXA-48 و ترکیبات فنازینی بررسی شد. در ادامه، نتایج انرژی اتصال و تعامل مولکولی بین آنزیم کارباپنماز OXA-48 و ترکیبات فنازین بررسی شدند، تا ترکیبی با توانایی مهاری بالقوه علیه آنزیم کارباپنماز OXA-48 شناسایی و به عنوان نامزد دارویی موثر برای بهرهبرداری بالینی معرفی گردد.
روش بررسی
انتخاب و آمادهسازی پروتئین: ساختار کریستالی آنزیم کارباپنماز OXA-48 با کد شناسایی PDB: 3HBR از بانک اطلاعات پروتئین (PDB) دریافت شد (11). در ادامه، نرمافزار AutoDock Tools4.2 برای آمادهسازی آنزیم کارباپنماز OXA-48 به عنوان گیرنده جهت مطالعات داکینگ مولکولی مورد استفاده قرار گرفت (12). به این ترتیب که مولکولهای آب، کوفاکتورها و سایر مولکولهایی که در جایگاه فعال گیرنده حضور ندارند، حذف شدند. اتمها با انواع اتمهای AutoDock تنظیم شده و سپس ترتیب پیوندها، اتمهای هیدروژن و بارهای Gasteiger-Marsili به ساختار پروتئین اضافه شدند. در نهایت، ساختار گیرنده در قالب PDBQT آماده شد (12). انتخاب و آمادهسازی جایگاههای اتصال آنزیم کارباپنمازOXA-48 برای انجام مطالعات داکینگ مولکولی به منظور تعیین ریشههای آمینواسیدی در جایگاه فعال آنزیم کارباپنماز OXA-48 که در انجام واکنش آنزیمی نقش دارند، فایل3HBR.pbb با استفاده از سرور PDBsum و نرمافزار LigPlot+ مورد مطالعه قرار گرفت (13). بر این اساس، ریشههای آمینواسیدی Ser118، Thr209، Arg214، Arg250، Ser70، KCX 73، Gln 124، Ile 102 و Ser244به عنوان جایگاههای اتصال برای برای اتصال انعطافپذیر انتخاب شدند. سپس نرمافزار AutoDock Tools4.2 برای پیش بینی جعبه شبکه در نقاط شبکه X = 26 Å، Y = 26 Å و Z = 26 Å استفاده شد و فاصله شبکه Å1 در نظر گرفته شد.
انتخاب و آماده سازی لیگاند: جهت انتخاب لیگاندها مقالات علمی مرتبط با متابولیتهای ثانویه فنازینها بررسی گردید. بر اساس متون معتبر علمی، متابولیتهای ثانویه فنازینها به دلیل ساختار شیمیایی منحصربهفرد دارای فعالیت بیولوژیکی و پتانسیل بالای اتصال به پروتئینهای هدف هستند و به دلیل ویژگیهای ضد میکروبی اثبات شدهشان، به عنوان لیگاندهای مناسب برای این مطالعه انتخاب شدند. اگرچه برخی از ترکیبات فنازینی پیشتر فعالیتهای زیستی از جمله اثرات ضد میکروبی نشان داده اند (14-17، 8)، اما هدف از انجام داکینگ مولکولی در این مطالعه، بررسی دقیقتر مکانیسم اتصال این ترکیبات به آنزیم کارباپنماز OXA-48 و درک مکانیسم عمل احتمالی این ترکیبات در سطح مولکولی و همچنین شناسایی ترکیباتی با پتانسیل مهارکنندگی بالاتر میباشد. بنابراین بر اساس مطالعات انجام شده، شش مولکول کوچک به نامهای Aotaphenazine، BaraphenazineD، PhenazinolinE، BaraphenazineE، PhenazinolinA و PhenazinolinD متعلق به خانواده فنازینها به عنوان لیگاند برای این مطالعه انتخاب شدند. ساختار دو بعدی این لیگاندها از پایگاه داده Pubchem (https://pubchem.ncbi.nlm.nih.gov/) با پسوند sdf دریافت شد و با استفاده از نرمافزارOpenBabel 2.3.2 به پسوند pdb تغییر پیدا کرد (18). بهینه سازی لیگاندها توسط نرمافزار Chem Draw(3D) (نسخه 17.1) انجام شد (https://perkinelmer-chemdraw-professional.software.informer.com/17.1). در نهایت ترکیبات بهینهسازی شده جهت انجام داکینگ مولکولی توسط نرمافزار Autodock tools به پسوند pdbqt تغییر پیدا کردند. نام و ساختار این ترکیبات در جدول 1 نشان داده شده است.
مطالعه داکینگ مولکولی: به منظور انجام داکینگ مولکولی از نرم افزار AutoDock Vina استفاده شد (19). به این صورت که داکینگ برای جایگاه اتصال تعریف شده آنزیم کارباپنماز OXA-48 به عنوان گیرنده و لیگاندهای آماده شده بهطور جداگانه انجام شد. انرژی تمایل لیگاندها به گیرنده به صورت کیلوکالری بر مول محاسبه شد، بهطوری که هرچه مقدار انرژی کمتر باشد تمایل لیگاند به گیرنده بیشتر است. نتایج اتصال و تعامل مولکولی بین آنزیم کارباپنماز OXA-48 و لیگاندها با استفاده از نرمافزار Pymol و LigPlot+ ارزیابی و مشاهده شد (13،20).
نتایج
تجزیه و تحلیل ساختار کارباپنماز OXA-48: ارزیابی ساختار آنزیم کارباپنماز OXA-48 نشان داد که این آنزیم یک هومودایمر با چهار زنجیره است که هر زنجیره 256 ریشهی آمینواسیدی دارد. جایگاه فعال این آنزیم در یک کانال باریک با عرض، عمق و طول 5، 10 و Å20 قرار گرفته است. این کانال توسط چهار ریشهی آمینواسیدی Arg214، Gln124، Ile102 و Ser244 احاطه شده است. بر اساس مقایسه با سایر بتالاکتامازهای کلاس D، ریشههای آمینواسیدی Ser70، Lys73 (لیزین کاربامیله شده) و Arg250 که در جایگاه فعال آنزیم قرار دارند، حفاظت شده هستند (شکل 1).
ارزیابی برهمکنش آنزیم کارباپنماز OXA-48 با فنازینها: اتصال مولکولی بین آنزیم کارباپنماز OXA-48 و فنازینها توسط Autodock Vina انجام شد. نتایج اتصال مولکولی نشان داد که تمام لیگاندها میل اتصال منفی به پروتئین هدف داشتند (جدول 2). محدودهی انرژی تمایل لیگاندها به گیرنده از 9/9- تا 4/9- کیلوکالری بر مول بود، به این صورت که بالاترین و کمترین انرژی تمایل به ترتیب مربوط به ترکیب Aotaphenazine و Phenazinolin D تعیین شد. ریشههای آمینواسیدی درگیر در پیوندهای هیدروژنی و غیر هیدروژنی با هرکدام از لیگاندها که از طریق بررسی نتایج داکینگ مولکولی توسط نرم افزار LigPlot+ تعیین شده با جزئیات بیشتر در جدول 2 و شکل 2 آورده شده است.
دارا بودن خصوصیات فیزیکوشیمیایی مطلوب در کنار اثربخشی دارویی از جمله شاخصهای مهم جهت ارزیابی یک مولکول به عنوان یک نامزد دارویی مناسب است. لذا در این مطالعه خصوصیات فیزیکوشیمیایی ترکیبات فنازینی انتخاب شده از طریق پایگاه داده PubChem (https://pubchem.ncbi.nlm.nih.gov/) بررسی گردید.

شکل 1: ساختار سه بعدی آنزیم کارباپنماز OXA-48 (با کد شناسایی: PDB: 3HBR)
جدول:1 نام و ساختار ترکیبات فنازین مورد استفاده در این مطالعه

جدول 2: ارزیابی انرژی اتصال (کیلوکالری بر مول) ترکیبات فنازین به آنزیم کارباپنماز OXA-48 و بررسی ریشههای آمینواسیدی درگیر در پیوندهای هیدروژنی و غیرهیدروژنی.


شکل 2: ساختار دو بعدی از برهمکنش آمینواسیدهای آنزیم کارباپنماز OXA-48 درگیر در پیوندهای هیدروژنی و غیرهیدروژنی با ترکیبات فنازینی. آمینواسیدهای درگیر در پیوندهای هیدروژنی با رنگ سیاه، آمینواسیدهای درگیر در پیوندهای غیر هیدروژنی با رنگ قرمز و طول پیوندهای هیدروژنی به صورت خط چین سبز رنگ نشان داده شده است. الف: Aotaphenazine، ب: BaraphenazineD، پ: PhenazinolinE، ت: BaraphenazineE، ث: Phenazinolin A، ج: Phenazinolin D
جدول 3: ارزیابی خواص فیزیکوشیمیایی ترکیبات فنارینها بر اساس نرمافزار OSIRIS Data Warrior

بحث
انتروباکتریاسههای مقاوم به کارباپنم اکنون به یک معضل جهانی در زمینه درمان عفونتهای این خانواده تبدیل شدهاند، زیرا دیگر به کارباپنمها که آخرین خط درمانی آنتیبیوتیکها در بسیاری از بیمارستانها هستند، حساسیت نشان نمیدهند. در حالیکه سفتازیدیم-آویباکتام نتایج امیدوارکنندهای در درمان این عفونتها نشان داده است، اما در بسیاری از کشورها به طور گسترده استفاده نمیشود و مقاومت در برابر این ترکیب نیز در حال افزایش است. با توجه به فوریت این وضعیت، سازمان بهداشت جهانی در به روز رسانی سال 2024 خود، این باکتریها را به عنوان پاتوژنهایی که نیازمند درمانهای جدید هستند، فهرست کرده است. درک مکانیسمهای مقاومت به کارباپنمها در انتروباکتریاسهها، مانند افزایش بیان پمپهای افلاکس، جهش در پورینها و تولید کارباپنمازها، برای یافتن داروهای جدید برای درمان آنها ضروری است. در میان کارباپنمازهای تولید شده توسط انتروباکتریاسهها، OXA-48 شایعترین کارباپنماز در منطقه خاورمیانه، از جمله ایران است. با اینحال، بهترین درمان برای عفونتهای ناشی از باکتریهای تولیدکننده کارباپنماز OXA-48 هنوز مشخص نیست. سفتازیدیم- آویباکتام یک درمان تایید شده برای انتروباکتریاسههای مقاوم به کارباپنم است. با اینحال، ظهور مقاومت در برابر این درمان نگران کننده است. وابربکتام یک مهارکننده بتالاکتاماز است که در ترکیب با مروپنم برای درمان عفونتهای ناشی از انتروباکتریاسههای مقاوم به کارباپنم استفاده می شود، اما وابربکتام در برابر کارباپنماز OXA-48 عملکردی ندارد (5). برای رسیدگی به این مسئله حیاتی، در این مطالعه از رویکرد غربالگری مجازی برای شناسایی مولکولهای کوچک از خانوادهی فنازینها با توانایی مهار کارباپنماز OXA-48 استفاده شد. فنازینها، متابولیتهای ثانویهای هستند که توسط باکتریهای مختلف از جمله استرپتومایسسها تولید میشوند و خواص ضدتوموری، ضد انگلی و ضدمالاریایی آنها تأیید شده است (8). بنایراین در این مطالعه به منظور یافتن مهارکنندههای جدید کارباپنماز OXA-48، ترکیبات فنازین (متابولیتهای ثانویه گونه های استرپتومایسس) انتخاب شده و سپس با روشهای داکینگ مولکولی نحوه اتصال و برهم کنشهای این آنزیم و ترکیبات بررسی گردید. نتیجه مطالعه حاضر نشان داد که ترکیبات فنازین مورد مطالعه، میتوانند با اتصال به جایگاه فعال آنزیم کارباپنماز OXA-48، موجب مهار این آنزیم شوند. بر اساس نتایج داکینگ، پتانسیل مهاری ترکیبات مورد بررسی با یکدیگر تفاوت داشته و قویترین اتصالات با بیشترین پتانسیل مهاری مربوط به ترکیباتAotaphenazine ، PhenazinolinE و BaraphenazineD است. ترکیب Aotaphenazine با انرژی اتصال 9/9- کیلوکالری بر مول و ازطریق سه پیوند هیدروژنی با آمینواسیدهای Ser70، Ser118 و Thr209و همچنین شش پیوند هیدروفوبی با آمینواسیدهایIle102 ،Trp105 ،Gly210 ،Tyr211 ، Thr213 و Arg250برهمکنش دارد. ترکیب PhenazinolinE با انرژی اتصال 9/6- کیلوکالری بر مول ازطریق دو پیوند هیدروژنی با آمینواسیدهای Ser118 و Thr213و همچنین هفت پیوند هیدروفوبی با آمینواسیدهای Trp105، Ile102، Arg100، Tyr117،Val120 ، Tyr211 و Arg214 برهمکنش دارد. در حالیکه ترکیب BaraphenazineD با انرژی اتصال 9/6- کیلوکالری بر مول ازطریق یک پیوند هیدروژنی با آمینواسید Thr209 و یازده پیوند هیدروفوبی با آمینواسیدهایIle102 ،Trp105 ،Met115 ،Tyr117 ،Lys116 ،Ser118 ، Met195،Leu196 ،Thr197 ، Lys208 و Ala194 برهمکنش دارد (جدول 1 و شکل 2). همچنین ترکیبات BaraphenazineE ، Phenazinolin A و Phenazinolin D انرژهای اتصال مناسبی را نشان دادند که به ترتیب شامل 9/5-، 9/4- و 9/4- کیلوکالری بر مول میباشند (جدول 1). در همه موارد، ترکیبات فنازینی توسط پیوندهای هیدروژنی با حداقل یکی از این آمینواسیدهای کلیدی Ser70، Ser118 و Thr209 اتصال و برهمکنش دارند. با این حال، Aotaphenazine، PhenazinolinE و Phenazinolin D پیوندهای هیدروژنی بیشتری نسبت به سایر ترکیبات انتخاب شده نشان دادند. علیرغم تعداد پیوندهای هیدروژنی، تعاملات هیدروفوبیک بیشترین تعاملات در کمپلکسها بودند. علاوه بر این، نتایج این مطالعه نشان داد که همه ترکیبات فنازینی انتخاب شده میتوانند به طور مؤثر با آمینواسیدهای جایگاه فعال کارباپنماز OXA-48 تعامل داشته باشند و در نتیجه میتوانند از طریق مسدود کردن جایگاه اتصال کارباپنماز OXA-48، در درمان عفونت باکتریایی به همراه آنتی بیوتیکها نقش داشته باشند. اثبات شده است که حدود 90٪ از ترکیبات فعال خوراکی از قانون لیپنسکی (RO5) پیروی میکنند. قانون لیپینسکی پیشبینی میکند که ترکیباتی که وزن مولکولی کمتر از 500 دالتون، فاکتور چربی دوستی کمتر از 5، تعداد اتمهای پذیرنده هیدروژن کمتر از 10 و تعداد اتمهای دهنده هیدروژن کمتر از 5 داشته باشند دارای ویژگیهای خوراکی بودن هستند (21). لذا برای بررسی ترکیبات از لحاظ این قانون در این مطالعه، خواص فیزیکوشیمیایی مهم ترکیبات انتخاب شده توسط نرمافزار OSIRIS Data Warrior بررسی شد (22) (جدول3). نتایج نشان داد که ترکیبات انتخاب شده از قانون لیپنسکی پیروی میکنند و قابلیت خوراکی بودن را دارند. با توجه به نیاز مقابله با گسترش جهانی مقاومت آنتیبیوتیکی، در این مطالعه ترکیبات اولیه معقولی برای بررسیهای تجربی در مهار آنزیم کارباپنماز OXA-48 شناسایی گردید که ممکن است برای محققانی که به شناسایی درمانهای جدید با متابولیتهای ثانویه علاقه مند هستند، قابلتوجه باشند.
نتیجهگیری
نتایج این مطالعه نشان میدهد که ترکیبات فنازینی دارای انرژی تمایل بالا و برهمکنشهای قابل توجهی با آنزیم کارباپنماز OXA-48 بوده و همچنین خصوصیات فیزیکوشیمیایی مناسب را از خود نشان میدهند. لذا این ترکیبات که به صورت طبیعی در باکتریهای جنس Streptomyces وجود دارند، میتوانند پتانسیل بالایی برای مهار آنزیم کارباپنماز OXA-48 و مقابله با مقاومت آنتی بیوتیکی داشته باشند. پیشنهاد میشود که اثر مهاری این ترکیبات در شرایط آزمایشگاهی و بالینی نیز بررسی شود تا اطمینان بیشتری از خاصیت ضدباکتریایی آنها به دست آید.
سپاسگزاری
این مطالعه برگرفته از طرح تحقیقاتی میباشد که در معاونت تحقیقات و فناوری دانشگاه علوم پزشکی تهران و معاونت تحقیقات و فناوری دانشگاه علوم پزشکی قم به ترتیب با کد 71572-101-1-1403 و 14031849 به ثبت رسیده است. بدین وسیله نویسندگان مقاله مراتب قدردانی و سپاس خود را از تمام افرادی که در این مطالعه همکاری داشته اند اعلام مینمایند.
حامی مالی: حامیان مالی این مطالعه دانشگاه علوم پزشکی تهران و دانشگاه علوم پزشکی قم
تعارض در منافع: وجود ندارد.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه علوم پزشکی تهران و دانشگاه علوم پزشکی قم تایید شده است (کد 1403.100. IR.TUMS.MEDICINE.REC.).
مشارکت نویسندگان
حوریه کلهر و مریم سیروسی در ارائه ایده حوریه کلهر و مریم سیروسی در طراحی مطالعه، حوریه کلهر و مریم سیروسی در جمعآوری دادهها، حوریه کلهر و مریم سیروسی در تجزیه و تحلیل دادهها مشارکت داشته و همه نویسندگان در تدوین، ویرایش اولیه و نهایی مقاله و پاسخگویی به سوالات مرتبط با مقاله سهیم هستند.
References:
1- Lomovskaya O, Sun D, Rubio-Aparicio D, Nelson K, Tsivkovski R, Griffith DC, Dudley MN. Vaborbactam: Spectrum of Beta-Lactamase Inhibition and Impact of Resistance Mechanisms on Activity in Enterobacteriaceae. Antim Agents and Chem 2017; 61(11): 01443-17.
2- Haidar G, Clancy CJ, Chen L, Samanta P, Shields RK, Kreiswirth BN, Nguyen MH. Identifying Spectra of Activity and Therapeutic Niches for Ceftazidime-Avibactam and Imipenem-Relebactam Against Carbapenem-Resistant Enterobacteriaceae. Antimicrobial Agents and Chemother 2017; 61(9): e00642-17.
3- Ambler RP, Coulson AF, Frère J-M, Ghuysen J-M, Joris B, Forsman M, et al. A Standard Numbering Scheme for the Class a Beta-Lactamases. Biochem J 1991; 276(Pt1): 269-70.
4- Touati A, Mairi A. Epidemiology of Carbapenemase-Producing Enterobacterales in the Middle East: A Systematic Review. Expert Rev Anti Infect Ther 2020; 18(3): 241-50.
5- Tompkins K, van Duin D. Treatment for Carbapenem-Resistant Enterobacterales Infections: Recent Advances and Future Directions. Eur J Clin Microbiol & Infect Dis 2021; 40(10): 2053-68.
6- Wang X, Abbas M, Zhang Y, Elshahawi SI, Ponomareva LV, Cui Z, et al. Baraphenazines A–G, Divergent Fused Phenazine-Based Metabolites From A Himalayan Streptomyces. J Nat Prod 2019; 82(6): 1686-93.
7- Huang W, Wan Y, Zhang S, Wang C, Zhang Z, Su H, et al. Recent Advances in Phenazine Natural Products: Chemical Structures and Biological Activities. Molecules 2024; 29(19): 4771.
8- Laursen JB, Nielsen J. Phenazine Natural Products: Biosynthesis, Synthetic Analogues, And Biological Activity. Chem Rev 2004; 104(3): 1663-86.
9- Prieto-Martínez FD, Arciniega M, Medina-Franco JL. Molecular Docking: Current Advances and Challenges. TIP Revista Especializada En Ciencias Químico-Biológicas 2018; 21: 1-23.
10- Asiamah I, Obiri SA, Tamekloe W, Armah FA, Borquaye LS. Applications of Molecular Docking in Natural Products-Based Drug Discovery. Scientific African 2023; 20: e01593.
11- Docquier J-D, Calderone V, De Luca F, Benvenuti M, Giuliani F, Bellucci L, et al. Crystal Structure of the OXA-48 Β-Lactamase Reveals Mechanistic Diversity Among Class D Carbapenemases. Chem Biol 2009; 16(5): 540-7.
12- Morris GM, Huey R, Lindstrom W, Sanner MF, Belew RK, Goodsell DS, Olson AJ. Autodock4 and Autodocktools4: Automated Docking with Selective Receptor Flexibility. J Comput Chem 2009; 30(16): 2785-91.
13- Laskowski RA, Swindells MB. Ligplot+: Multiple Ligand–Protein Interaction Diagrams for Drug Discovery. ACS Publications; 2011; 51(12): 2778-86.
14- Sousa CA, Ribeiro M, Vale F, Simões M. Phenazines: Natural Products for Microbial Growth Control. HLife 2024; 2(3): 100-12.
15- Guttenberger N, Blankenfeldt W, Breinbauer R. Recent Developments in the Isolation, Biological Function, Biosynthesis, and Synthesis of Phenazine Natural Products. Bioorganic & Medicinal Chemistry 2017; 25(22): 6149-66.
16- Pierson LS, Pierson EA. Metabolism and Function of Phenazines in Bacteria: Impacts on the Behavior of Bacteria in the Environment and Biotechnological Processes. Appl Microbiol Biotechnol 2010; 86(6): 1659-70.
17- Hernandez ME, Kappler A, Newman DK. Phenazines and other Redox-Active Antibiotics Promote Microbial Mineral Reduction. Appl Environ Microbiol 2004; 70(2): 921-8.
18- O'Boyle NM, Banck M, James CA, Morley C, Vandermeersch T, Hutchison GR. Open Babel: An Open Chemical Toolbox. J Cheminform 2011; 3: 33.
19- Trott O, Olson AJ. Autodock Vina: Improving The Speed and Accuracy of Docking with a New Scoring Function, Efficient Optimization, and Multithreading. J Comput Chem 2010; 31(2): 455-61.
20- DeLano WL. Pymol: An Open-Source Molecular Graphics Tool. CCP4 Newsl Protein Crystallogr 2002; 40(1): 82-92.
21- Lipinski CA. Drug-Like Properties and the Causes of Poor Solubility and Poor Permeability. J pharmacological and toxicological methods 2000; 44(1): 235-49.
22- Sander T, Freyss J, Korff Mv, R ufener C. Datawarrior: An Open-Source Program for Chemistry Aware Data Visualization and Analysis. J Chem Inform Model 2015; 55(2): 460-73.
1- Lomovskaya O, Sun D, Rubio-Aparicio D, Nelson K, Tsivkovski R, Griffith DC, Dudley MN. Vaborbactam: Spectrum of Beta-Lactamase Inhibition and Impact of Resistance Mechanisms on Activity in Enterobacteriaceae. Antim Agents and Chem 2017; 61(11): 01443-17.
2- Haidar G, Clancy CJ, Chen L, Samanta P, Shields RK, Kreiswirth BN, Nguyen MH. Identifying Spectra of Activity and Therapeutic Niches for Ceftazidime-Avibactam and Imipenem-Relebactam Against Carbapenem-Resistant Enterobacteriaceae. Antimicrobial Agents and Chemother 2017; 61(9): e00642-17.
3- Ambler RP, Coulson AF, Frère J-M, Ghuysen J-M, Joris B, Forsman M, et al. A Standard Numbering Scheme for the Class a Beta-Lactamases. Biochem J 1991; 276(Pt1): 269-70.
4- Touati A, Mairi A. Epidemiology of Carbapenemase-Producing Enterobacterales in the Middle East: A Systematic Review. Expert Rev Anti Infect Ther 2020; 18(3): 241-50.
5- Tompkins K, van Duin D. Treatment for Carbapenem-Resistant Enterobacterales Infections: Recent Advances and Future Directions. Eur J Clin Microbiol & Infect Dis 2021; 40(10): 2053-68.
6- Wang X, Abbas M, Zhang Y, Elshahawi SI, Ponomareva LV, Cui Z, et al. Baraphenazines A–G, Divergent Fused Phenazine-Based Metabolites From A Himalayan Streptomyces. J Nat Prod 2019; 82(6): 1686-93.
7- Huang W, Wan Y, Zhang S, Wang C, Zhang Z, Su H, et al. Recent Advances in Phenazine Natural Products: Chemical Structures and Biological Activities. Molecules 2024; 29(19): 4771.
8- Laursen JB, Nielsen J. Phenazine Natural Products: Biosynthesis, Synthetic Analogues, And Biological Activity. Chem Rev 2004; 104(3): 1663-86.
9- Prieto-Martínez FD, Arciniega M, Medina-Franco JL. Molecular Docking: Current Advances and Challenges. TIP Revista Especializada En Ciencias Químico-Biológicas 2018; 21: 1-23.
10- Asiamah I, Obiri SA, Tamekloe W, Armah FA, Borquaye LS. Applications of Molecular Docking in Natural Products-Based Drug Discovery. Scientific African 2023; 20: e01593.
11- Docquier J-D, Calderone V, De Luca F, Benvenuti M, Giuliani F, Bellucci L, et al. Crystal Structure of the OXA-48 Β-Lactamase Reveals Mechanistic Diversity Among Class D Carbapenemases. Chem Biol 2009; 16(5): 540-7.
12- Morris GM, Huey R, Lindstrom W, Sanner MF, Belew RK, Goodsell DS, Olson AJ. Autodock4 and Autodocktools4: Automated Docking with Selective Receptor Flexibility. J Comput Chem 2009; 30(16): 2785-91.
13- Laskowski RA, Swindells MB. Ligplot+: Multiple Ligand–Protein Interaction Diagrams for Drug Discovery. ACS Publications; 2011; 51(12): 2778-86.
14- Sousa CA, Ribeiro M, Vale F, Simões M. Phenazines: Natural Products for Microbial Growth Control. HLife 2024; 2(3): 100-12.
15- Guttenberger N, Blankenfeldt W, Breinbauer R. Recent Developments in the Isolation, Biological Function, Biosynthesis, and Synthesis of Phenazine Natural Products. Bioorganic & Medicinal Chemistry 2017; 25(22): 6149-66.
16- Pierson LS, Pierson EA. Metabolism and Function of Phenazines in Bacteria: Impacts on the Behavior of Bacteria in the Environment and Biotechnological Processes. Appl Microbiol Biotechnol 2010; 86(6): 1659-70.
17- Hernandez ME, Kappler A, Newman DK. Phenazines and other Redox-Active Antibiotics Promote Microbial Mineral Reduction. Appl Environ Microbiol 2004; 70(2): 921-8.
18- O'Boyle NM, Banck M, James CA, Morley C, Vandermeersch T, Hutchison GR. Open Babel: An Open Chemical Toolbox. J Cheminform 2011; 3: 33.
19- Trott O, Olson AJ. Autodock Vina: Improving The Speed and Accuracy of Docking with a New Scoring Function, Efficient Optimization, and Multithreading. J Comput Chem 2010; 31(2): 455-61.
20- DeLano WL. Pymol: An Open-Source Molecular Graphics Tool. CCP4 Newsl Protein Crystallogr 2002; 40(1): 82-92.
21- Lipinski CA. Drug-Like Properties and the Causes of Poor Solubility and Poor Permeability. J pharmacological and toxicological methods 2000; 44(1): 235-49.
22- Sander T, Freyss J, Korff Mv, R ufener C. Datawarrior: An Open-Source Program for Chemistry Aware Data Visualization and Analysis. J Chem Inform Model 2015; 55(2): 460-73.
نوع مطالعه: پژوهشي |
موضوع مقاله:
میکروبیولوژی
دریافت: 1403/11/9 | پذیرش: 1403/12/19 | انتشار: 1404/6/15
دریافت: 1403/11/9 | پذیرش: 1403/12/19 | انتشار: 1404/6/15
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |







