دوره 30، شماره 10 - ( دی 1401 )
جلد 30 شماره 10 صفحات 5299-5290 |
برگشت به فهرست نسخه ها
Ethics code: IR.IAU.KAU.REC.1400.142
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Alipour M, Moghanibashi M, Naeimi S. Bioinformatics Identification of miRNA-mRNA Regulatory Network Contributing Primary Lung Cancer. JSSU 2023; 30 (10) :5290-5299
URL: http://jssu.ssu.ac.ir/article-1-5711-fa.html
URL: http://jssu.ssu.ac.ir/article-1-5711-fa.html
علیپور مرضیه، مغنیباشی مهدی، نعیمی سیروس. شناسایی بیوانفورماتیکی شبکه تنظیمی miRNA-mRNA مرتبط با سرطان ریه اولیه. مجله علمي پژوهشي دانشگاه علوم پزشكي شهید صدوقی يزد. 1401; 30 (10) :5290-5299
متن کامل [PDF 970 kb]
(672 دریافت)
| چکیده (HTML) (1740 مشاهده)
References:
1- Higuchi R, Goto T, Nakagomi T, Hirotsu Y, Oyama T, Amemiya K, Mochizuki H, Omata M. Discrimination Between Primary Lung Cancer and Lung Metastases by Genomic Profiling. JTO Clin Res Rep 2021; 2(12):100255.
2- Barta JA, Powell CA, Wisnivesky JP. Global Epidemiology of Lung Cancer. Ann Glob Health 2019; 85(1): 8.
3- Jones GS, Baldwin DR. Recent Advances in the Management of Lung Cancer. Clin Med (Lond) 2018; 18(2): 41-46.
4- Baran K, Brzeziańska-Lasota E. Proteomic Biomarkers of Non-Small Cell Lung Cancer Patients. Adv Respir Med 2021; 89: 419-26.
5- Li MY, Liu LZ, Dong M. Progress on Pivotal Role and Application of Exosome in Lung Cancer Carcinogenesis, Diagnosis, Therapy and Prognosis. Mol Cancer 2021; 20(1): 22.
6- Chen K, Sun J, Zhao H, Jiang R, Zheng J, Li Z, et al. Non-Invasive Lung Cancer Diagnosis and Prognosis Based on Multi-Analyte Liquid Biopsy. Mol Cancer 2021; 20(1): 23.
7- Goossens N, Nakagawa S, Sun X, Hoshida Y. Cancer Biomarker Discovery and Validation. Transl Cancer Res 2015; 4(3): 256-69.
8- Jiang HG, Dai CH, Xu YP, Jiang Q, Xia XB, Shu Y, et al. Four Plasma Mirnas Act as Biomarkers for Diagnosis and Prognosis of Non-Small Cell Lung Cancer. Oncol Lett 2021; 22(5): 792.
9- Yi M, Liao Z, Deng L, Xu L, Tan Y, Liu K, et al. High Diagnostic Value of Mirnas for NSCLC: Quantitative Analysis for Both Single and Combined Mirnas In Lung Cancer. Ann Med 2021; 53(1): 2178-93.
10- Han W, Cui H, Liang J, Su X. Role of Microrna-30c in Cancer Progression. J Cancer 2020; 11(9): 2593-601.
11- Zhang J, Duy Le T, Liu L, He J, Li J. Identifying Mirna Synergistic Regulatory Networks in Heterogeneous Human Data Via Network Motifs. Mol Biosyst 2016; 12(2): 454-63.
12- Van Der Sijde F, Homs MYV, Van Bekkum ML, Van Den Bosch TPP, Bosscha K, Besselink MG, et al. Serum Mir-373-3p and Mir-194-5p are Associated with Early Tumor Progression During FOLFIRINOX Treatment in Pancreatic Cancer Patients: A Prospective Multicenter Study. Int J Mol Sci 2021; 22(20): 10902.
13- Jing SY, Jing SQ, Liu LL, Xu LF, Zhang F, Gao JL. Down-Expression of Mir-373 Predicts Poor Prognosis of Glioma and Could be a Potential Therapeutic Target. Eur Rev Med Pharmacol Sci 2017; 21(10): 2421-25.
14- Shah JA, Khattak S, Rauf MA, Cai Y, Jin J. Potential Biomarkers of Mir-371-373 Gene Cluster in Tumorigenesis. Life (Basel) 2021; 11(9): 984.
15- Chen W, Lin G, Yao Y, Chen J, Shui H, Yang Q, Wang X, Et Al. Microrna Hsa-Let-7e-5p As A Potential Prognosis Marker For Rectal Carcinoma With Liver Metastases. Oncol Lett 2018; 15(5): 6913-24.
16- He RQ, Wu PR, Xiang XL, Yang X, Liang HW, Qiu XH, Et Al. Downregulated Mir-23b-3p Expression Acts as a Predictor of Hepatocellular Carcinoma Progression: A Study Based on Public Data and RT-Qpcr Verification. Int J Mol Med 2018; 41(5): 2813-31.
17- Begum S, Hayashi M, Ogawa T, Jabboure FJ, Brait M, Izumchenko E, Et Al. An Integrated Genome-Wide Approach To Discover Deregulated Micrornas in Non-Small Cell Lung Cancer: Clinical Significance Of Mir-23b-3p Deregulation. Sci Rep 2015; 5: 13236.
18- Moya L, Meijer J, Schubert S, Matin F, Batra J. Assessment of Mir-98-5p, Mir-152-3p, Mir-326 and Mir-4289 Expression as Biomarker for Prostate Cancer Diagnosis. Int J Mol Sci 2019; 20(5): 1154.
19- Rana S, Valbuena GN, Curry E, Bevan CL, Keun HC. Micrornas as Biomarkers for Prostate Cancer Prognosis: A Systematic Review and a Systematic Reanalysis of Public Data. Br J Cancer 2022; 126(3): 502-13.
20- Lorena Quirico, Francesca Orso. The Power of Micrornas as Diagnostic and Prognostic Biomarkers in Liquid Biopsies. Cancer Drug Resistance 2020; 3(2): 117-39.
21- Bryant R, Pawlowski T, Catto JWF, Marsden G, Vessella RL, Rhees B, et al. Changes in Circulating Microrna Levels Associated with Prostate Cancer. British journal of cancer 2012; 106(4); 768-74.
22- Kong W, Cheng Y, Liang H, Chen Q, Xiao C, Li K, Huang Z, Zhang J. Prognostic Value of Mir-17-5p in Cancers: A Meta-Analysis. Onco Targets Ther 2018; 11: 3541-49.
23- Hasakova K, Reis R, Vician M, Zeman M, Herichova I. Expression of Mir-34a-5p is Up-Regulated in Human Colorectal Cancer and Correlates with Survival and Clock Gene PER2 Expression. Plos One 2019; 14(10): E0224396.
24- Ma R, Wang C, Wang J, Wang D, Xu J. Mirna–Mrna Interaction Network in Non-Small Cell Lung Cancer. Interdisciplinary Sciences: Computational Life Sciences 2016; 8(3): 209-19.
25- Subat S, Inamura K, Ninomiya H, Nagano H, Okumura S, Ishikawa Y. Unique Microrna and Mrna Interactions in EGFR-Mutated Lung Adenocarcinoma. JCM 2018; 7(11): 419.
26- Nie R, Niu W, Tang T, Zhang J, Zhang X. Integrating Microrna Expression, Mirna-Mrna Regulation Network and Signal Pathway: A Novel Strategy for Lung Cancer Biomarker Discovery. Peerj 2021; 9: E12369.
27- Li DS, Ainiwaer JL, Sheyhiding I, Zhang Z, Zhang LW. Identification of Key Long Non-Coding Rnas as Competing Endogenous Rnas for Mirna-Mrna in Lung Adenocarcinoma. Eur Rev Med Pharmacol Sci 2016; 20(11): 2285-95.
28- Liang L, Zhang L, Zhang J, Bai S, Fu H. Identification of Circrna-Mirna-Mrna Networks for Exploring the Fundamental Mechanism in Lung Adenocarcinoma. Oncotargets Ther 2020; 13: 2945-55.
29- Cai X, Lin L, Zhang Q, Wu W, Su A. Bioinformatics Analysis of the Circrna–Mirna–Mrna Network for Non-Small Cell Lung Cancer. J Int Med Res 2020; 48(6): 0300060520929167.
30- Wang XJ, Gao J, Wang Z, Yu Q. Identification of a Potentially Functional Microrna–Mrna Regulatory Network in Lung Adenocarcinoma Using a Bioinformatics Analysis. Front Cell Deve Biol 2021; 9, 641840.
31- Wang J, Yu XF, Ouyang N, Zhao S, Yao H, Guan X, et al. Microrna and Mrna Interaction Network Regulates the Malignant Transformation of Human Bronchial Epithelial Cells Induced by Cigarette Smoke. Front in Oncol 2019; 9: 1029.
32- Zhang W, Zhang Q, Che L, Xie Z, Cai X, Gong L, et al. Using Biological Information to Analyze Potential Mirna-Mrna Regulatory Networks in the Plasma of Patients with Non-Small Cell Lung Cancer. BMC Cancer 2022; 22(1): 299.
33- Sheervalilou R, Shahraki J, Shahraki O, Shirvalilou S, Ghaznavi H. A Review on Micrornas’ Function, Detection and Evaluation Methods, Expression Dysregulation Mechanisms and Possible Applications in Clinical Phase as Diagnostic, Prognostic and Therapeutic Biomarkers of Lung Cancer Patients. RJMS 2020; 27(3):101-21.
متن کامل: (1830 مشاهده)
مقدمه
در سالهای اخیر، افزایش تعداد بیماران مبتلا به سرطان ریه، آن را به شایعترین سرطان تشخیص داده شده در زنان و مردان و علت اصلی مرگ و میر ناشی از سرطان در سراسر جهان تبدیل کرده است (1،2). سرطان ریه بیماری با پیشآگهی نامطلوب (3) و نرخ بقای پنج ساله بسیار پایین (13- 6 درصد) میباشد که دو زیر گروه اصلی؛ کارسینومای سلول کوچک ریه (SCLC) و کارسینومای سلول غیر کوچک ریه (NSCLC) را شامل میشود. با وجود تلاشهای گسترده سه دهه گذشته در تشخیص سریع بیماران مبتلا به این بدخیمی، 50 درصد از بیماران NSCLC، به عنوان شایعترین نوع این سرطان از لحاظ بافت شناسی (شامل 85 درصد از کل سرطانهای ریه)، در زمان متاستاز شناسایی میگردند، چراکه همچنان تمایز تومورهای تهاجمی از تومورهای اولیه ریه در اقدامات بالینی چالش برانگیز است (6-4). با تکمیل پروژه ژنوم انسان و پیشرفت تکنیکهای دقیق توالییابی، اخیراً نشانگرهای زیستی مبتنی بر ژنوم، در سیستم مرحلهبندی تومورهای مختلف بهکار گرفته شدهاند. بهنظر میرسد شناسایی نشانگرهای ژنومی جدیدی که قادرند حضور تومورها در مراحل اولیه را تشخیص دهند و عود سرطان را پیشبینی کنند، میتواند ابزار مفیدی را برای تشخیص زودهنگام NSCLC با پتانسیل کاهش مرگ و میر فراهم کند (7،8). در میان تمام نشانگرهای زیستی تحت بررسی، microRNAها (miRNAs) به عنوان دستهای از RNAهای کوچک غیر کد کننده و دارای نقش مهم در تنظیم بیان ژن، یکی از امیدوارکنندهترین موارد از نظر تشخیص زودهنگام NSCLC در نظر گرفته میشوند (9). MicroRNAها (miRNAs یا miRs) RNAهای تک رشتهای درونزاد غیر کدکننده با طول 18 تا 24 نوکلئوتید عمدتا واقع در نواحی اینترونی و رونوشتهای غیر کد کننده ژنهای رمزشونده میباشند که با اتصال به ناحیه 3′ UTR از ملکول mRNA هدف خود، ترجمه mRNA را پس از رونویسی مهار میکنند. در سالهای اخیر، مطالعات متعددی ثابت کردهاند که miRNAها بهصورت منفرد در قالب انکوژنها یا ژنهای سرکوب کننده تومور، تکثیر، تمایز، تهاجم و فرآیند متاستاز سلول¬های سرطانی را تنظیم مینمایند (10). علاوه بر این، یک miRNA میتواند یک یا چند mRNA را هدف قرار دهد و همراه با mRNA هدف خود در یک شبکه تنظیمی نقش همافزایی در پیشرفت سرطان داشته باشد (11). لذا هماکنون با توجه به در دسترس بودن حجم زیادی از دادههای بهدستآمده از تکنیک هایی با توان عملیاتی بالا و پیشرفت الگوریتمها و ابزارهای بیوانفورماتیکی، امکان شناسایی miRNAهای دخیل در پیشرفت سرطان و بررسی روابط بین آنها و mRNAهای هدفشان در شبکه های تنظیمی حاصل شده است تا بتوان از آنها به عنوان کاندیدای نشانگرهای زیستی مهم برای کمک به تشخیص و پیشآگهی استفاده کرد (10،11). از اینرو، این مطالعه با هدف شناسایی شبکه تنظیمی miRNA-mRNA مرتبط با سرطان NSCLC اولیه با رویکرد بیوانفورماتیکی انجام شد.
روش بررسی
در مطالعه حاضر، از پایگاه داده اطلس ژنوم سرطانTCGA (The Cancer Genome Atlas ) (https://cancergenome.nih.gov/) پروفایل بیان ژنی 320 بیمار سرطان اولیه سلولهای غیر کوچک ریه به عنوان گروه مورد و 49 فرد سالم به عنوان گروه شاهد استخراج شد. سپس با استفاده از بسته نرمافزار TCGAbiolinks و نرمافزار آماری Rآنالیزهای بعدی انجام گرفت. در ادامه، پیشپردازشهای اولیه برای داده¬های ترانسکریپتومی شامل؛ حذف ژنهایی با بیان صفر یا نزدیک به صفر با معیار CMP (Cladistic Maximum Parsimony) کمتر از 10 در 50 درصد نمونهها با استفاده از پکیج edgeR در نرمافزارR انجام گرفت. نرمالسازی دادهها بر اساس روش TMM (TheTrimmed Mean of the M-values) و انتقال دادهها به حالت لگاریتمی بر پایه 2 توسط پکیج limma صورت گرفت. برای محاسبه تفاوت بیان بین گروههای مورد مقایسه یعنی نمونههای بافت سرطان ریه اولیه و کنترلهای سالم از روش linear model استفاده شد و محاسبه سطح معنیداری بین گروهها از طریق multiple hypothesis testing انجام گرفت. در تمامی آنالیزها معیار FDR<0.05 (False Discovery Rate) به عنوان معیار انتخاب mRNAها و miRNAهای دارای تغییر بیان بکار گرفته شد. نهایتاً از ماتریکس پروفایل بیانی بهدست آمده جهت انجام آنالیزهای بعدی مطالعه استفاده شد. متعاقباً برای mRNAهای اختصاصی شناسایی شده، با استفاده از پایگاه TargetScan (http://www.targetscan.org/) و miRWalk (http://mirwalk.umm.uni-heidelberg.de/)، که در آنها همبستگی بین miRNAها و ژنهای هدف از طریق آزمایشات تایید شده است، miRNAهایی که میتوانند آنها را هدف قرار دهند شناسایی شدند. miRNAهای پیشبینی شده توسط هر دو الگوریتم برای mRNAهای بهدست آمده، در نظر گرفته شدند. نهایتاً شبکه تنظیمکننده تعامل miRNA-mRNA با استفاده از نرمافزار Cytoscape (V 4) به تصویر کشیده شد.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه آزاد اسلامی واحد کازرون تصویب شده است (کد اخلاق IR.IAU.KAU.REC.1400.142).
نتایج
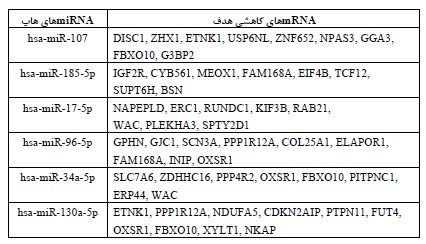
تحلیل بیواتفورماتیکی mRNAها و miRNAهای دارای تفاوت بیان بین نمونههای بافت سرطان ریه اولیه و کنترلهای سالم، با استفاده از پکیجهای edgeR و limma، مجموع 748 mRNAاختصاصی با افزایش بیان در نمونههای سرطان اولیه سلولهای غیر کوچک ریه را نشان داد که تعداد زیادی از آنها در شبکه تعاملی توسط 7 miRNA شامل؛ hsa-miR-373-3p، hsa-let-7a-5p، hsa-miR-23b-3p، hsa-miR-152-3p، hsa-miR-216a-3p، hsa-miR-106-5p و hsa-let-7i-5p تنظیم میگردند (شکل 1). با توجه به نقش miRNAهای هاب شناسایی شده در تنظیم ژنهای اختصاصی سرطان NSCLC اولیه، میتوان با بهگیری آنها در عملیات تشخیصی بالینی، حضور تومور NSCLC را در مراحل اولیه تشخیص داد. miRNAهای هاب تنظیم کننده mRNAهای دچار افزایش بیان درجدول 1 به همراه اهداف ژنی آنها فهرست شدهاند. همچنین در بررسی شبکه تعاملی miRNA-mRNA دخیل در سرطان سلولهای غیر کوچک ریه اولیه، 6 miRNA هاب شامل؛ has-miR-107، has-miR-17-5p، has-185-5p، has-miR-34a-5p، has-miR-130a-5p و has-96-5p (شکل 2) به عنوان تنظیم کنندههای اصلی تعداد زیادی از 355 mRNA اختصاصی شناسایی شده در نمونههای سرطان اولیه سلول های غیر کوچک ریه با بیان کاهش یافته، در نظر گرفته شدند. miRNAهای هاب تنظیم کننده mRNAهای دچار کاهش بیان در جدول 2 به همراه اهداف ژنی آنها فهرست شده اند.
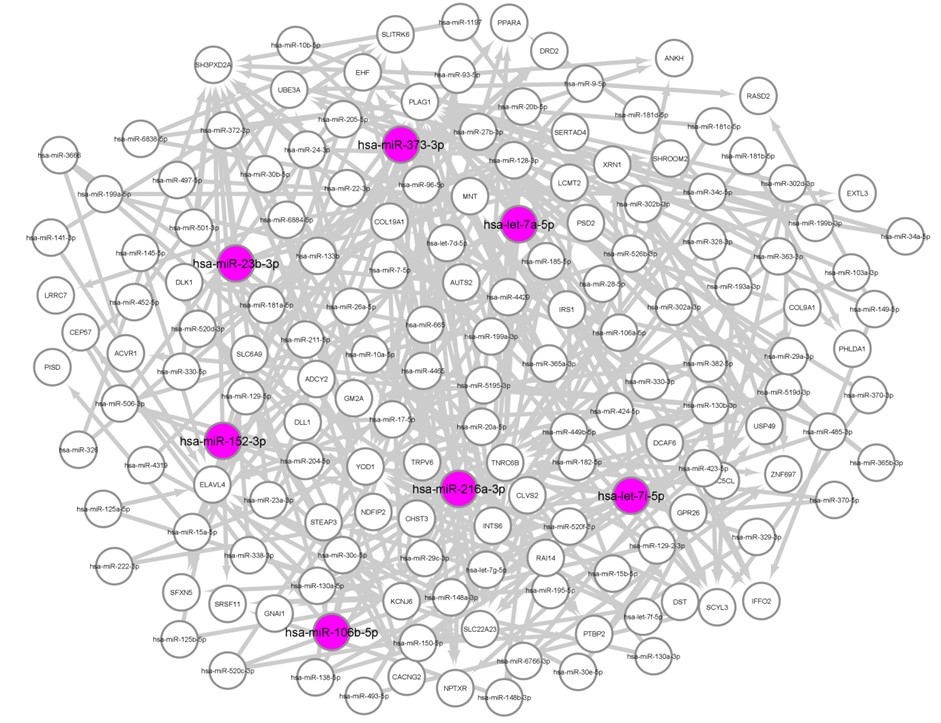
شکل 1: شبکه تعاملی miRNA-mRNA دخیل در سرطان سلول های غیر کوچک ریه اولیه.
نقاط رنگی نشان دهنده miRNAهای هاب تنظیمکننده mRNAهای دچار افزایش بیان در بافت سرطانی NSCLC اولیه میباشد.
جدول 1. لیست miRNAها هاب و mRNAهای افزایشی هدف در بافت سرطانی NSCLC اولیه
.JPG)
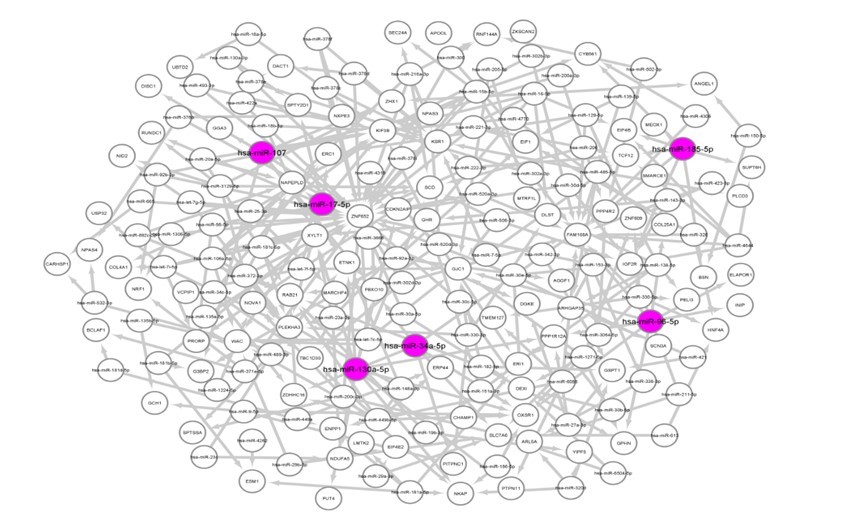
شکل 2: شبکه تعاملی miRNA-mRNA دخیل در سرطان سلولهای غیر کوچک ریه اولیه.
نقاط رنگی نشان دهنده miRNAهای هاب تنظیم کننده mRNAهای دچار کاهش بیان در بافت سرطانی NSCLC اولیه می باشد.
جدول 2: لیست miRNAها هاب و mRNAهای کاهشی هدف در بافت سرطانی NSCLC اولیه
بحث
نتایج مطالعه حاضر، مبنی بر شناسایی 7 miRNA تنظیمکننده ژن های اختصاصی دچار افزایش بیان در نمونههای سرطان اولیه سلول های غیر کوچک ریه، شامل؛ hsa-miR-373-3p، hsa-let-7a-5p، hsa-miR-23b-3p، hsa-miR-152-3p، hsa-miR-216a-3p، hsa-miR-106-5p و hsa-let-7i-5p و 6 miRNA تنظیم کننده ژن های اختصاصی با بیان کاهش یافته در این وضعیت سرطانی، شامل؛ has-miR-107، has-miR-17-5p، has-185-5p، has-miR-34a-5p، has-miR-130a-5p و has-96-5p، در شبکه های تنظیمی miRNA-mRNA مرتبط با NSCLC اولیه، بر توانایی این miRNAها در تمایز تومورهای اولیه از تومورهای متاستازی ریه تاکید دارد و نوید استفاده از آنها به عنوان ابزاری برای تشخیص زودرس و پیشآگهی مناسب سرطان ریه را میدهد. مطابق با نتایج ما، ون در و همکاران hsa-miR-373-3p را به عنوان مارکر پیشبینی کننده پیشرفت تومور اولیه آدنوکارسینومای داکتال پانکراس معرفی کردهاند (12). در مطالعه دیگری نیز hsa-miR-373-3p فاکتور پیشبینی کننده بالقوهای برای پیشآگهی گلیوما درنظر گرفته شده است (13). همچنین علیشاه و همکاران نقش hsa-miR-373-3p را به عنوان یک مارکر ملکولی در تشخیص و پیشگری از سرطانهای مختلف گزارش کردهاند (14). در مطالعه چن و همکاران hsa-let-7a-5p مارکر پیشآگهی بالقوه برای کارسینومای رکتال در نظر گرفته شده است (15). hsa-miR-23b-3p به عنوان مارکر پیشبینی کننده پیشرفت سرطان کارسینومای هپاتوسلولار اولیه اعلام شده است (16). بهطور مشابه در مطالعه بیگم و همکاران miR-23b-3p مارکر بالقوهای برای پیشآگهی NSCLC در مراحل اولیه گزارش شده است (17). در مطالعه مویا و رانا hsa-miR-152-3p به ترتیب یک ابزار تشخیصی و یک مارکر پیشآگهی موثر برای سرطان پروستات گزارش شده است (18،19). اهمیت hsa-miR-106-5p نیز به عنوان مارکر تشخیصی و پیشآگهی در سرطان کولورکتال اثبات شده است (20). اهمیت گروه دیگر از miRNAهای شناسایی شده در این پژوهش به عنوان بیومارکرهای تشخیصی سرطان نیز در مطالعات سال¬های اخیر گزارش شده است. به عنوان مثال در سال 2021، has-miR-107 مارکری بالقوه برای تشخیص و پیش آگهی سرطان پروستات شناخته شده است (21). در سال 2018، miR-17-5p بهعنوان مارکری برای نظارت بر توسعه و پیشرفت سرطان توصیه شده است (22). has-miR-34a-5p نیز در سال 2019، بهعنوان یک مارکر پیش آگهی برای مراحل اولیه سرطان کولورکتال در بیمارانی با جنسیت مذکر مطرح شده است (23). ما و همکاران در سال 2016 از طریق چندین نرم افزار بیوانفورماتیکی به بررسی ارتباط میان miRNA–mRNA در سرطان سلول غیر کوچک ریه پرداختند نتایج آنها نشان داد که سه miRNA از جمله:miR-1207-5p, miR-1228* miR-939 در تنظیم بسیاری از ژنها از جمله: ST8SIA2, MED1, HDAC4, SPN, نقش دارد (24). شبات و همکاران در سال 2018 با استفاده از ریز آرایهها به بررسی پروفایل بیان miRNA – mRNA در آدنوکارسینومای ریه پرداختند تجزیه و تحلیل دادهها نشان داد miR-532-3p ,miR-500a-3p, miR-224-5p ,miR-502-3p ,miR-532-5p بهعنوان miRNAهای مختص جهش EGFR هستند (25). نای و همکارانش در سال 2021 با استفاده از پایگاه داده TCGAو GEOبه بررسی ارتباط miRNA-mRNAمرتبط با NSCLC پرداختند در این مطالعه miR-708-5p به عنوان نشانگر زیستی شناسایی شد (26). لی و همکاران در سال 2016 با استفاده از نرمافزارهای بیوانفورماتیکی به بررسی کنش miRNA-mRNA , miRNA-lncRNA در آدنوکارسینومای ریه پرداختند تجزیه و تحلیل آنها نشان داد MEG3 ,MIAT ,LINC00115 میتوانند بهعنوان اهداف درمانی آدنوکارسینومای ریه باشند (27). لیانگ و همکاران در سال 2020 با استفاده از نرمافزار بیوانفورماتیک به بررسی نقش circRNA-miRNA-mRNA در آدنوکارسینوم ریه پرداختند نتایج آنها نشان داد پنج circRNAs از جمله: hsa_circ_0072088, hsa_circ 0082564, hsa_circ_0008274, hsa_circ_0000519 , hsa_circ_0003528 بهطور متفاوت بیان شدهاند که میتواند به عنوان اهداف درمانی قرار گیرند (28). سای و همکاران در سال 2020 به شناسایی ژنهای هاب پرداختند نتایج نشان داد ده ژن هاب، بیانDTL و RRM2را تحت تاثیر قرار میدهد و در پیشآگهی بیماران با NSCLC مرتبط است در نتیجه مولکولهای شناسایی شده از جمله: hsa_circ_0001947/ hsa-miR-637/ RRM2 و hsa_circ_0072305/ hsa-miR-127-5p/ DTL در بروز و توسعه NSCLC دخیل است (29). وانگ و همکاران در سال2021 با استفاده از ابزارهای بیوانفورماتیکی به بررسی شبکه تنظیمی microRNA–mRNA در آدنوکارسینومای ریه پرداختند در این مطالعه پنج microRNA از جمله: miR-539-5p, miR-656-3p, miR-2110, let-7b-5p, and miR-92b-3p به عنوان microRNAهای هاب شناسایی شدند (30). وانگ و همکاران در سال 2019 با استفاده از پایگاه داده TCGA به بررسی شبکه تنظیمی miRNAs – mRNAs در سلولهای اپیتلیال برونش انسان پرداختند نتایج آنها نشان داد کهmiR-96-5p, miR-93-5p, miR-106-5p, miR-190a-5p, miR-195-5p, miR-1-3p با سرطان ریه مرتبط هستند (31). زهنگ و همکاران در سال 2020 با استفاده از پایگاه داده GEO و TCGA به تجزیه و تحلیل بیان و پیشآگهی ژنهای هاب در NSCLC پرداختند نتایج آنها نشان داد بیانhsa-miR-199a-5p , hsa-miR-186-5p در خون محیطی بیماران NSCLC بیشتر از افراد سالم است (32). با توجه به نتایج حاصل شده از مرور جامع شبکههای ملکولی miRNA-mRNA مرتبط با سرطان ریه اولیه که در این مطالعه صورت گرفته است، میتوان miRNAها را در بهروزرسانی روشهای تشخیص بیماران NSCLC اولیه موثر دانست، چراکه دیدگاههای نوین مولکولی قادرند به بهبود کیفیت زندگی بیماران کمک کنند و به عنوان رویکرهای کاملاً مستقل برای غربالگری، شناسایی و درمان مطرح شوند (33).
نتیجهگیری
درک بیشتر از بیولوژی و ژنتیک سرطان ریه در پی انجام بررسیهای مولکولی میتواند به شناخت نشانگرهای زیستی در راستای غربالگری، بهبود پیشآگهی، تسریع در توسعه اهداف درمانی جدید یا بهکارگیری روشهای درمانی موثر در مراحل اولیه بیماری منجر گردد. در این بین شناسایی پروفایل مولکولی miRNAهای دارای تغییر بیان به عنوان عامل موثر بر انحراف مکانیسمهای تنظیم بیان ژن میتواند رویکردی مناسب برای تشخیص، طبقه¬بندی و درمان هدفمند سرطان پیچیده و ناهمگن ریه باشد.
سپاسگزاری
این مقاله برگرفته از پایاننامه مقطع دکتری تخصصی نویسنده اول مصوب دانشگاه آزاد اسلامی واحد کازرون در سال 1400 میباشد. نویسـندگان بـر خـود لازم مـیداننـد از آقای محمد مهدور و کلیه عزیزانی که نهایت همکاری را در اجرای این پژوهش داشته¬اند، کمال تشکر و قدردانی را به عمل آورند.
حامی مالی: ندارد.
تعارض در منافع: وجود ندارد.
در سالهای اخیر، افزایش تعداد بیماران مبتلا به سرطان ریه، آن را به شایعترین سرطان تشخیص داده شده در زنان و مردان و علت اصلی مرگ و میر ناشی از سرطان در سراسر جهان تبدیل کرده است (1،2). سرطان ریه بیماری با پیشآگهی نامطلوب (3) و نرخ بقای پنج ساله بسیار پایین (13- 6 درصد) میباشد که دو زیر گروه اصلی؛ کارسینومای سلول کوچک ریه (SCLC) و کارسینومای سلول غیر کوچک ریه (NSCLC) را شامل میشود. با وجود تلاشهای گسترده سه دهه گذشته در تشخیص سریع بیماران مبتلا به این بدخیمی، 50 درصد از بیماران NSCLC، به عنوان شایعترین نوع این سرطان از لحاظ بافت شناسی (شامل 85 درصد از کل سرطانهای ریه)، در زمان متاستاز شناسایی میگردند، چراکه همچنان تمایز تومورهای تهاجمی از تومورهای اولیه ریه در اقدامات بالینی چالش برانگیز است (6-4). با تکمیل پروژه ژنوم انسان و پیشرفت تکنیکهای دقیق توالییابی، اخیراً نشانگرهای زیستی مبتنی بر ژنوم، در سیستم مرحلهبندی تومورهای مختلف بهکار گرفته شدهاند. بهنظر میرسد شناسایی نشانگرهای ژنومی جدیدی که قادرند حضور تومورها در مراحل اولیه را تشخیص دهند و عود سرطان را پیشبینی کنند، میتواند ابزار مفیدی را برای تشخیص زودهنگام NSCLC با پتانسیل کاهش مرگ و میر فراهم کند (7،8). در میان تمام نشانگرهای زیستی تحت بررسی، microRNAها (miRNAs) به عنوان دستهای از RNAهای کوچک غیر کد کننده و دارای نقش مهم در تنظیم بیان ژن، یکی از امیدوارکنندهترین موارد از نظر تشخیص زودهنگام NSCLC در نظر گرفته میشوند (9). MicroRNAها (miRNAs یا miRs) RNAهای تک رشتهای درونزاد غیر کدکننده با طول 18 تا 24 نوکلئوتید عمدتا واقع در نواحی اینترونی و رونوشتهای غیر کد کننده ژنهای رمزشونده میباشند که با اتصال به ناحیه 3′ UTR از ملکول mRNA هدف خود، ترجمه mRNA را پس از رونویسی مهار میکنند. در سالهای اخیر، مطالعات متعددی ثابت کردهاند که miRNAها بهصورت منفرد در قالب انکوژنها یا ژنهای سرکوب کننده تومور، تکثیر، تمایز، تهاجم و فرآیند متاستاز سلول¬های سرطانی را تنظیم مینمایند (10). علاوه بر این، یک miRNA میتواند یک یا چند mRNA را هدف قرار دهد و همراه با mRNA هدف خود در یک شبکه تنظیمی نقش همافزایی در پیشرفت سرطان داشته باشد (11). لذا هماکنون با توجه به در دسترس بودن حجم زیادی از دادههای بهدستآمده از تکنیک هایی با توان عملیاتی بالا و پیشرفت الگوریتمها و ابزارهای بیوانفورماتیکی، امکان شناسایی miRNAهای دخیل در پیشرفت سرطان و بررسی روابط بین آنها و mRNAهای هدفشان در شبکه های تنظیمی حاصل شده است تا بتوان از آنها به عنوان کاندیدای نشانگرهای زیستی مهم برای کمک به تشخیص و پیشآگهی استفاده کرد (10،11). از اینرو، این مطالعه با هدف شناسایی شبکه تنظیمی miRNA-mRNA مرتبط با سرطان NSCLC اولیه با رویکرد بیوانفورماتیکی انجام شد.
روش بررسی
در مطالعه حاضر، از پایگاه داده اطلس ژنوم سرطانTCGA (The Cancer Genome Atlas ) (https://cancergenome.nih.gov/) پروفایل بیان ژنی 320 بیمار سرطان اولیه سلولهای غیر کوچک ریه به عنوان گروه مورد و 49 فرد سالم به عنوان گروه شاهد استخراج شد. سپس با استفاده از بسته نرمافزار TCGAbiolinks و نرمافزار آماری Rآنالیزهای بعدی انجام گرفت. در ادامه، پیشپردازشهای اولیه برای داده¬های ترانسکریپتومی شامل؛ حذف ژنهایی با بیان صفر یا نزدیک به صفر با معیار CMP (Cladistic Maximum Parsimony) کمتر از 10 در 50 درصد نمونهها با استفاده از پکیج edgeR در نرمافزارR انجام گرفت. نرمالسازی دادهها بر اساس روش TMM (TheTrimmed Mean of the M-values) و انتقال دادهها به حالت لگاریتمی بر پایه 2 توسط پکیج limma صورت گرفت. برای محاسبه تفاوت بیان بین گروههای مورد مقایسه یعنی نمونههای بافت سرطان ریه اولیه و کنترلهای سالم از روش linear model استفاده شد و محاسبه سطح معنیداری بین گروهها از طریق multiple hypothesis testing انجام گرفت. در تمامی آنالیزها معیار FDR<0.05 (False Discovery Rate) به عنوان معیار انتخاب mRNAها و miRNAهای دارای تغییر بیان بکار گرفته شد. نهایتاً از ماتریکس پروفایل بیانی بهدست آمده جهت انجام آنالیزهای بعدی مطالعه استفاده شد. متعاقباً برای mRNAهای اختصاصی شناسایی شده، با استفاده از پایگاه TargetScan (http://www.targetscan.org/) و miRWalk (http://mirwalk.umm.uni-heidelberg.de/)، که در آنها همبستگی بین miRNAها و ژنهای هدف از طریق آزمایشات تایید شده است، miRNAهایی که میتوانند آنها را هدف قرار دهند شناسایی شدند. miRNAهای پیشبینی شده توسط هر دو الگوریتم برای mRNAهای بهدست آمده، در نظر گرفته شدند. نهایتاً شبکه تنظیمکننده تعامل miRNA-mRNA با استفاده از نرمافزار Cytoscape (V 4) به تصویر کشیده شد.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه آزاد اسلامی واحد کازرون تصویب شده است (کد اخلاق IR.IAU.KAU.REC.1400.142).
نتایج
تحلیل بیواتفورماتیکی mRNAها و miRNAهای دارای تفاوت بیان بین نمونههای بافت سرطان ریه اولیه و کنترلهای سالم، با استفاده از پکیجهای edgeR و limma، مجموع 748 mRNAاختصاصی با افزایش بیان در نمونههای سرطان اولیه سلولهای غیر کوچک ریه را نشان داد که تعداد زیادی از آنها در شبکه تعاملی توسط 7 miRNA شامل؛ hsa-miR-373-3p، hsa-let-7a-5p، hsa-miR-23b-3p، hsa-miR-152-3p، hsa-miR-216a-3p، hsa-miR-106-5p و hsa-let-7i-5p تنظیم میگردند (شکل 1). با توجه به نقش miRNAهای هاب شناسایی شده در تنظیم ژنهای اختصاصی سرطان NSCLC اولیه، میتوان با بهگیری آنها در عملیات تشخیصی بالینی، حضور تومور NSCLC را در مراحل اولیه تشخیص داد. miRNAهای هاب تنظیم کننده mRNAهای دچار افزایش بیان درجدول 1 به همراه اهداف ژنی آنها فهرست شدهاند. همچنین در بررسی شبکه تعاملی miRNA-mRNA دخیل در سرطان سلولهای غیر کوچک ریه اولیه، 6 miRNA هاب شامل؛ has-miR-107، has-miR-17-5p، has-185-5p، has-miR-34a-5p، has-miR-130a-5p و has-96-5p (شکل 2) به عنوان تنظیم کنندههای اصلی تعداد زیادی از 355 mRNA اختصاصی شناسایی شده در نمونههای سرطان اولیه سلول های غیر کوچک ریه با بیان کاهش یافته، در نظر گرفته شدند. miRNAهای هاب تنظیم کننده mRNAهای دچار کاهش بیان در جدول 2 به همراه اهداف ژنی آنها فهرست شده اند.

شکل 1: شبکه تعاملی miRNA-mRNA دخیل در سرطان سلول های غیر کوچک ریه اولیه.
نقاط رنگی نشان دهنده miRNAهای هاب تنظیمکننده mRNAهای دچار افزایش بیان در بافت سرطانی NSCLC اولیه میباشد.
جدول 1. لیست miRNAها هاب و mRNAهای افزایشی هدف در بافت سرطانی NSCLC اولیه
.JPG)

شکل 2: شبکه تعاملی miRNA-mRNA دخیل در سرطان سلولهای غیر کوچک ریه اولیه.
نقاط رنگی نشان دهنده miRNAهای هاب تنظیم کننده mRNAهای دچار کاهش بیان در بافت سرطانی NSCLC اولیه می باشد.
جدول 2: لیست miRNAها هاب و mRNAهای کاهشی هدف در بافت سرطانی NSCLC اولیه

بحث
نتایج مطالعه حاضر، مبنی بر شناسایی 7 miRNA تنظیمکننده ژن های اختصاصی دچار افزایش بیان در نمونههای سرطان اولیه سلول های غیر کوچک ریه، شامل؛ hsa-miR-373-3p، hsa-let-7a-5p، hsa-miR-23b-3p، hsa-miR-152-3p، hsa-miR-216a-3p، hsa-miR-106-5p و hsa-let-7i-5p و 6 miRNA تنظیم کننده ژن های اختصاصی با بیان کاهش یافته در این وضعیت سرطانی، شامل؛ has-miR-107، has-miR-17-5p، has-185-5p، has-miR-34a-5p، has-miR-130a-5p و has-96-5p، در شبکه های تنظیمی miRNA-mRNA مرتبط با NSCLC اولیه، بر توانایی این miRNAها در تمایز تومورهای اولیه از تومورهای متاستازی ریه تاکید دارد و نوید استفاده از آنها به عنوان ابزاری برای تشخیص زودرس و پیشآگهی مناسب سرطان ریه را میدهد. مطابق با نتایج ما، ون در و همکاران hsa-miR-373-3p را به عنوان مارکر پیشبینی کننده پیشرفت تومور اولیه آدنوکارسینومای داکتال پانکراس معرفی کردهاند (12). در مطالعه دیگری نیز hsa-miR-373-3p فاکتور پیشبینی کننده بالقوهای برای پیشآگهی گلیوما درنظر گرفته شده است (13). همچنین علیشاه و همکاران نقش hsa-miR-373-3p را به عنوان یک مارکر ملکولی در تشخیص و پیشگری از سرطانهای مختلف گزارش کردهاند (14). در مطالعه چن و همکاران hsa-let-7a-5p مارکر پیشآگهی بالقوه برای کارسینومای رکتال در نظر گرفته شده است (15). hsa-miR-23b-3p به عنوان مارکر پیشبینی کننده پیشرفت سرطان کارسینومای هپاتوسلولار اولیه اعلام شده است (16). بهطور مشابه در مطالعه بیگم و همکاران miR-23b-3p مارکر بالقوهای برای پیشآگهی NSCLC در مراحل اولیه گزارش شده است (17). در مطالعه مویا و رانا hsa-miR-152-3p به ترتیب یک ابزار تشخیصی و یک مارکر پیشآگهی موثر برای سرطان پروستات گزارش شده است (18،19). اهمیت hsa-miR-106-5p نیز به عنوان مارکر تشخیصی و پیشآگهی در سرطان کولورکتال اثبات شده است (20). اهمیت گروه دیگر از miRNAهای شناسایی شده در این پژوهش به عنوان بیومارکرهای تشخیصی سرطان نیز در مطالعات سال¬های اخیر گزارش شده است. به عنوان مثال در سال 2021، has-miR-107 مارکری بالقوه برای تشخیص و پیش آگهی سرطان پروستات شناخته شده است (21). در سال 2018، miR-17-5p بهعنوان مارکری برای نظارت بر توسعه و پیشرفت سرطان توصیه شده است (22). has-miR-34a-5p نیز در سال 2019، بهعنوان یک مارکر پیش آگهی برای مراحل اولیه سرطان کولورکتال در بیمارانی با جنسیت مذکر مطرح شده است (23). ما و همکاران در سال 2016 از طریق چندین نرم افزار بیوانفورماتیکی به بررسی ارتباط میان miRNA–mRNA در سرطان سلول غیر کوچک ریه پرداختند نتایج آنها نشان داد که سه miRNA از جمله:miR-1207-5p, miR-1228* miR-939 در تنظیم بسیاری از ژنها از جمله: ST8SIA2, MED1, HDAC4, SPN, نقش دارد (24). شبات و همکاران در سال 2018 با استفاده از ریز آرایهها به بررسی پروفایل بیان miRNA – mRNA در آدنوکارسینومای ریه پرداختند تجزیه و تحلیل دادهها نشان داد miR-532-3p ,miR-500a-3p, miR-224-5p ,miR-502-3p ,miR-532-5p بهعنوان miRNAهای مختص جهش EGFR هستند (25). نای و همکارانش در سال 2021 با استفاده از پایگاه داده TCGAو GEOبه بررسی ارتباط miRNA-mRNAمرتبط با NSCLC پرداختند در این مطالعه miR-708-5p به عنوان نشانگر زیستی شناسایی شد (26). لی و همکاران در سال 2016 با استفاده از نرمافزارهای بیوانفورماتیکی به بررسی کنش miRNA-mRNA , miRNA-lncRNA در آدنوکارسینومای ریه پرداختند تجزیه و تحلیل آنها نشان داد MEG3 ,MIAT ,LINC00115 میتوانند بهعنوان اهداف درمانی آدنوکارسینومای ریه باشند (27). لیانگ و همکاران در سال 2020 با استفاده از نرمافزار بیوانفورماتیک به بررسی نقش circRNA-miRNA-mRNA در آدنوکارسینوم ریه پرداختند نتایج آنها نشان داد پنج circRNAs از جمله: hsa_circ_0072088, hsa_circ 0082564, hsa_circ_0008274, hsa_circ_0000519 , hsa_circ_0003528 بهطور متفاوت بیان شدهاند که میتواند به عنوان اهداف درمانی قرار گیرند (28). سای و همکاران در سال 2020 به شناسایی ژنهای هاب پرداختند نتایج نشان داد ده ژن هاب، بیانDTL و RRM2را تحت تاثیر قرار میدهد و در پیشآگهی بیماران با NSCLC مرتبط است در نتیجه مولکولهای شناسایی شده از جمله: hsa_circ_0001947/ hsa-miR-637/ RRM2 و hsa_circ_0072305/ hsa-miR-127-5p/ DTL در بروز و توسعه NSCLC دخیل است (29). وانگ و همکاران در سال2021 با استفاده از ابزارهای بیوانفورماتیکی به بررسی شبکه تنظیمی microRNA–mRNA در آدنوکارسینومای ریه پرداختند در این مطالعه پنج microRNA از جمله: miR-539-5p, miR-656-3p, miR-2110, let-7b-5p, and miR-92b-3p به عنوان microRNAهای هاب شناسایی شدند (30). وانگ و همکاران در سال 2019 با استفاده از پایگاه داده TCGA به بررسی شبکه تنظیمی miRNAs – mRNAs در سلولهای اپیتلیال برونش انسان پرداختند نتایج آنها نشان داد کهmiR-96-5p, miR-93-5p, miR-106-5p, miR-190a-5p, miR-195-5p, miR-1-3p با سرطان ریه مرتبط هستند (31). زهنگ و همکاران در سال 2020 با استفاده از پایگاه داده GEO و TCGA به تجزیه و تحلیل بیان و پیشآگهی ژنهای هاب در NSCLC پرداختند نتایج آنها نشان داد بیانhsa-miR-199a-5p , hsa-miR-186-5p در خون محیطی بیماران NSCLC بیشتر از افراد سالم است (32). با توجه به نتایج حاصل شده از مرور جامع شبکههای ملکولی miRNA-mRNA مرتبط با سرطان ریه اولیه که در این مطالعه صورت گرفته است، میتوان miRNAها را در بهروزرسانی روشهای تشخیص بیماران NSCLC اولیه موثر دانست، چراکه دیدگاههای نوین مولکولی قادرند به بهبود کیفیت زندگی بیماران کمک کنند و به عنوان رویکرهای کاملاً مستقل برای غربالگری، شناسایی و درمان مطرح شوند (33).
نتیجهگیری
درک بیشتر از بیولوژی و ژنتیک سرطان ریه در پی انجام بررسیهای مولکولی میتواند به شناخت نشانگرهای زیستی در راستای غربالگری، بهبود پیشآگهی، تسریع در توسعه اهداف درمانی جدید یا بهکارگیری روشهای درمانی موثر در مراحل اولیه بیماری منجر گردد. در این بین شناسایی پروفایل مولکولی miRNAهای دارای تغییر بیان به عنوان عامل موثر بر انحراف مکانیسمهای تنظیم بیان ژن میتواند رویکردی مناسب برای تشخیص، طبقه¬بندی و درمان هدفمند سرطان پیچیده و ناهمگن ریه باشد.
سپاسگزاری
این مقاله برگرفته از پایاننامه مقطع دکتری تخصصی نویسنده اول مصوب دانشگاه آزاد اسلامی واحد کازرون در سال 1400 میباشد. نویسـندگان بـر خـود لازم مـیداننـد از آقای محمد مهدور و کلیه عزیزانی که نهایت همکاری را در اجرای این پژوهش داشته¬اند، کمال تشکر و قدردانی را به عمل آورند.
حامی مالی: ندارد.
تعارض در منافع: وجود ندارد.
References:
1- Higuchi R, Goto T, Nakagomi T, Hirotsu Y, Oyama T, Amemiya K, Mochizuki H, Omata M. Discrimination Between Primary Lung Cancer and Lung Metastases by Genomic Profiling. JTO Clin Res Rep 2021; 2(12):100255.
2- Barta JA, Powell CA, Wisnivesky JP. Global Epidemiology of Lung Cancer. Ann Glob Health 2019; 85(1): 8.
3- Jones GS, Baldwin DR. Recent Advances in the Management of Lung Cancer. Clin Med (Lond) 2018; 18(2): 41-46.
4- Baran K, Brzeziańska-Lasota E. Proteomic Biomarkers of Non-Small Cell Lung Cancer Patients. Adv Respir Med 2021; 89: 419-26.
5- Li MY, Liu LZ, Dong M. Progress on Pivotal Role and Application of Exosome in Lung Cancer Carcinogenesis, Diagnosis, Therapy and Prognosis. Mol Cancer 2021; 20(1): 22.
6- Chen K, Sun J, Zhao H, Jiang R, Zheng J, Li Z, et al. Non-Invasive Lung Cancer Diagnosis and Prognosis Based on Multi-Analyte Liquid Biopsy. Mol Cancer 2021; 20(1): 23.
7- Goossens N, Nakagawa S, Sun X, Hoshida Y. Cancer Biomarker Discovery and Validation. Transl Cancer Res 2015; 4(3): 256-69.
8- Jiang HG, Dai CH, Xu YP, Jiang Q, Xia XB, Shu Y, et al. Four Plasma Mirnas Act as Biomarkers for Diagnosis and Prognosis of Non-Small Cell Lung Cancer. Oncol Lett 2021; 22(5): 792.
9- Yi M, Liao Z, Deng L, Xu L, Tan Y, Liu K, et al. High Diagnostic Value of Mirnas for NSCLC: Quantitative Analysis for Both Single and Combined Mirnas In Lung Cancer. Ann Med 2021; 53(1): 2178-93.
10- Han W, Cui H, Liang J, Su X. Role of Microrna-30c in Cancer Progression. J Cancer 2020; 11(9): 2593-601.
11- Zhang J, Duy Le T, Liu L, He J, Li J. Identifying Mirna Synergistic Regulatory Networks in Heterogeneous Human Data Via Network Motifs. Mol Biosyst 2016; 12(2): 454-63.
12- Van Der Sijde F, Homs MYV, Van Bekkum ML, Van Den Bosch TPP, Bosscha K, Besselink MG, et al. Serum Mir-373-3p and Mir-194-5p are Associated with Early Tumor Progression During FOLFIRINOX Treatment in Pancreatic Cancer Patients: A Prospective Multicenter Study. Int J Mol Sci 2021; 22(20): 10902.
13- Jing SY, Jing SQ, Liu LL, Xu LF, Zhang F, Gao JL. Down-Expression of Mir-373 Predicts Poor Prognosis of Glioma and Could be a Potential Therapeutic Target. Eur Rev Med Pharmacol Sci 2017; 21(10): 2421-25.
14- Shah JA, Khattak S, Rauf MA, Cai Y, Jin J. Potential Biomarkers of Mir-371-373 Gene Cluster in Tumorigenesis. Life (Basel) 2021; 11(9): 984.
15- Chen W, Lin G, Yao Y, Chen J, Shui H, Yang Q, Wang X, Et Al. Microrna Hsa-Let-7e-5p As A Potential Prognosis Marker For Rectal Carcinoma With Liver Metastases. Oncol Lett 2018; 15(5): 6913-24.
16- He RQ, Wu PR, Xiang XL, Yang X, Liang HW, Qiu XH, Et Al. Downregulated Mir-23b-3p Expression Acts as a Predictor of Hepatocellular Carcinoma Progression: A Study Based on Public Data and RT-Qpcr Verification. Int J Mol Med 2018; 41(5): 2813-31.
17- Begum S, Hayashi M, Ogawa T, Jabboure FJ, Brait M, Izumchenko E, Et Al. An Integrated Genome-Wide Approach To Discover Deregulated Micrornas in Non-Small Cell Lung Cancer: Clinical Significance Of Mir-23b-3p Deregulation. Sci Rep 2015; 5: 13236.
18- Moya L, Meijer J, Schubert S, Matin F, Batra J. Assessment of Mir-98-5p, Mir-152-3p, Mir-326 and Mir-4289 Expression as Biomarker for Prostate Cancer Diagnosis. Int J Mol Sci 2019; 20(5): 1154.
19- Rana S, Valbuena GN, Curry E, Bevan CL, Keun HC. Micrornas as Biomarkers for Prostate Cancer Prognosis: A Systematic Review and a Systematic Reanalysis of Public Data. Br J Cancer 2022; 126(3): 502-13.
20- Lorena Quirico, Francesca Orso. The Power of Micrornas as Diagnostic and Prognostic Biomarkers in Liquid Biopsies. Cancer Drug Resistance 2020; 3(2): 117-39.
21- Bryant R, Pawlowski T, Catto JWF, Marsden G, Vessella RL, Rhees B, et al. Changes in Circulating Microrna Levels Associated with Prostate Cancer. British journal of cancer 2012; 106(4); 768-74.
22- Kong W, Cheng Y, Liang H, Chen Q, Xiao C, Li K, Huang Z, Zhang J. Prognostic Value of Mir-17-5p in Cancers: A Meta-Analysis. Onco Targets Ther 2018; 11: 3541-49.
23- Hasakova K, Reis R, Vician M, Zeman M, Herichova I. Expression of Mir-34a-5p is Up-Regulated in Human Colorectal Cancer and Correlates with Survival and Clock Gene PER2 Expression. Plos One 2019; 14(10): E0224396.
24- Ma R, Wang C, Wang J, Wang D, Xu J. Mirna–Mrna Interaction Network in Non-Small Cell Lung Cancer. Interdisciplinary Sciences: Computational Life Sciences 2016; 8(3): 209-19.
25- Subat S, Inamura K, Ninomiya H, Nagano H, Okumura S, Ishikawa Y. Unique Microrna and Mrna Interactions in EGFR-Mutated Lung Adenocarcinoma. JCM 2018; 7(11): 419.
26- Nie R, Niu W, Tang T, Zhang J, Zhang X. Integrating Microrna Expression, Mirna-Mrna Regulation Network and Signal Pathway: A Novel Strategy for Lung Cancer Biomarker Discovery. Peerj 2021; 9: E12369.
27- Li DS, Ainiwaer JL, Sheyhiding I, Zhang Z, Zhang LW. Identification of Key Long Non-Coding Rnas as Competing Endogenous Rnas for Mirna-Mrna in Lung Adenocarcinoma. Eur Rev Med Pharmacol Sci 2016; 20(11): 2285-95.
28- Liang L, Zhang L, Zhang J, Bai S, Fu H. Identification of Circrna-Mirna-Mrna Networks for Exploring the Fundamental Mechanism in Lung Adenocarcinoma. Oncotargets Ther 2020; 13: 2945-55.
29- Cai X, Lin L, Zhang Q, Wu W, Su A. Bioinformatics Analysis of the Circrna–Mirna–Mrna Network for Non-Small Cell Lung Cancer. J Int Med Res 2020; 48(6): 0300060520929167.
30- Wang XJ, Gao J, Wang Z, Yu Q. Identification of a Potentially Functional Microrna–Mrna Regulatory Network in Lung Adenocarcinoma Using a Bioinformatics Analysis. Front Cell Deve Biol 2021; 9, 641840.
31- Wang J, Yu XF, Ouyang N, Zhao S, Yao H, Guan X, et al. Microrna and Mrna Interaction Network Regulates the Malignant Transformation of Human Bronchial Epithelial Cells Induced by Cigarette Smoke. Front in Oncol 2019; 9: 1029.
32- Zhang W, Zhang Q, Che L, Xie Z, Cai X, Gong L, et al. Using Biological Information to Analyze Potential Mirna-Mrna Regulatory Networks in the Plasma of Patients with Non-Small Cell Lung Cancer. BMC Cancer 2022; 22(1): 299.
33- Sheervalilou R, Shahraki J, Shahraki O, Shirvalilou S, Ghaznavi H. A Review on Micrornas’ Function, Detection and Evaluation Methods, Expression Dysregulation Mechanisms and Possible Applications in Clinical Phase as Diagnostic, Prognostic and Therapeutic Biomarkers of Lung Cancer Patients. RJMS 2020; 27(3):101-21.
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |







