دوره 28، شماره 10 - ( دی 1399 )
جلد 28 شماره 10 صفحات 3133-3121 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Nabavizadeh N, Hojati Z. Exploring the Correlation between TNC Gene and Osteoporosis. JSSU 2020; 28 (10) :3121-3133
URL: http://jssu.ssu.ac.ir/article-1-5183-fa.html
URL: http://jssu.ssu.ac.ir/article-1-5183-fa.html
نبوی زاده نسرین السادات، حجتی زهره. بررسی ارتباط بین ژنTNC و بیماری پوکی استخوان. مجله علمي پژوهشي دانشگاه علوم پزشكي شهید صدوقی يزد. 1399; 28 (10) :3121-3133
متن کامل [PDF 927 kb]
(1030 دریافت)
| چکیده (HTML) (2627 مشاهده)
متن کامل: (2401 مشاهده)
مقدمه
پوکی استخوان بهعنوان یکی از بیماریهای مهم قرن بیست و یکم در نظر گرفته میشود و تقریباً 200 میلیون نفر در سطح جهان به این بیماری مبتلا هستند که تأثیر قابل توجهی در میزان مرگ و میر دارد (1). عمدتاً پوکی استخوان با کاهش تراکم مواد معدنی استخوان (BMD) Bone mineral density و افزایش خطر شکنندگی و وخیمتر شدن وضعیت استخوان همراه است و متأسفانه این اپیدمی ویرانگر بهطور خاموش پیش میرود تا زمانی که یک شکستگی رخ دهد (2). با توجه بهارتباط نزدیک بین روند پیری و پاتوژنز پوکی استخوان، تحقیقات در مورد مکانیسمهای از بین رفتن استخوان همراه با افزایش سن در سالهای اخیر بهطور قابل توجهی گسترش یافته است که شامل ترکیبی از مطالعات بالینی و مولکولی است. بهطور کلی، پیری یک فرایند پیچیده است که در اثر مجموعهای از فاکتورهای محیطی و ژنتیکی روی میدهد. در روند پیری، مجموعهای از تغییرات زیانآور در سلولها و بافتها انباشته میشوند که در نهایت منجر به افزایش خطر بیماری و مرگ میگردند (3). در مقابل پیری و بیماریهای رایج مثل دیابت، چاقی و بسیاری از انواع سرطانها که معمولاً در اثر مجموعهای از علائم متعدد ژنتیکی و فاکتورهای محیطی ایجاد میشوند، بیماریهای نادر وجود دارند که معمولاً در اثر جهش در یک ژن ایجاد میشوند و اثبات روند بیماریزایی آن سادهتر است. کشف مکانیسمهای دخیل در بیماریهای نادر میتواند به فهم عمیقتر ما از بیماریهای رایج مرتبط و مکانیسمهای مولکولی آنها کمک کند. برای مثال گرچه بیماران مبتلا به بیماری نادر و وراثتی پوکی استخوان درصد بسیار کمی از جمعیّت را تشکیل میدهند، امّا فرایند پوکی استخوان با افزایش سن بهصورت رایج در زندگی بسیاری از انسانها وجود دارد و تلاشهای بسیار زیادی تاکنون برای پیشگیری و درمان آن صورت گرفته است. مطالعه بر روی بیماری نادر پوکی استخوان یک ابزار بسیار مفید برای مطالعه اختصاصی روند پوکی استخوان در دوران پیری است بدین صورت که ژنهای دخیل در بیماری و مکانیسم آنها میتوانند در طراحی بسیاری از درمانها مورد استفاده قرار گیرند. از جمله بیماریهای ارثی و مونوژنیک شناخته شده در ارتباط با پوکی استخوان میتوان به استئوژنزیز ایمپرفکتا (osteogenesis imperfecta)، سندرم بروک (Bruck syndrome)، استئوپتروزیس (osteopetrosis)، سندرم استئوپورزیس-سودوگلیوما (osteoporosis-pseudoglioma syndrome)، بیماری ون بوخم (von Buchem disease)، اسکلروستئوزتئوزیز (sclerosteosteosis)، استئولیز خانوادگی (familial osteolysis)، بیماری پاژه (Paget’s disease)، هیپوفسفاتازی (hypophosphatasia)، هایپرپاراتیروئیدیسم نوزادی(neonatal hyperparathyroidism) و پیکنودیزوستوز (pyknodysostosis) اشاره کرد (4). در این مطالعه از نمونههای استئوژنزیز ایمپرفکتا استفاده گردید که در آن بیماران از بدو تولد از پوکی استخوان شدید و شکستگیهای متعدد استخوانی رنج می¬برند.
همچنین ژن (TNC) Tenascin C به عنوان ژن کاندید در روند پوکیاستخوان انتخاب شد. ژنTNC یک گلیکوپروتئین ماتریکس خارج سلولی را کد میکند که در طی رشد استخوان و مورفوژنز توسط سلولهای استئوبلاست ساخته میشود (6،5). بررسیها نشان داده است که TNC در تمایز استخوانی استئوبلاست و معدنی شدن بافت استخوان نقش دارد. در طی این تمایز، ممکن است TNC به عنوان یک واسطه برای تشکیل استخوان جدید توسط سیگنال TGF-β فعال شود (8، 7). علاوه بر این، پروتئین (BMP) Bone morphogenetic protein و فاکتورهای رشد Wnt یا فشار مکانیکی و استرس میتوانند بیان TNC را در استئوبلاستها افزایش دهند (10، 9). هدف اصلی این مطالعه بررسی ارتباط ژن TNC با بیماری پوکی استخوان با استفاده از روشهای بیوانفورماتیکی و آزمایشگاهی است. لازم بهذکر است که این مطالعه به منظور کاندید کردن بیومارکر پوکی استخوان صورت گرفته است و بهدلیل تعداد بسیار محدود نمونههای بیماران مبتلا بهاستئوژنزیز ایمپرفکتا به عنوان مدل مونوژن و نادر پوکی استخوان، لازم است این نتایج در مطالعات گستردهتر با جامعه آماری مناسب متشکل از بیماران مبتلا به انواع پوکی استخوان و افراد سالم تأیید نهایی شود.
روش بررسی
مطالعات بیوانفورماتیک و زیست شناسی سامانهها
به منظور به دست آوردن برهمکنشهای پروتئینی TNC از پایگاه داده زیستی String (https://string-db.org/) استفاده گردید. این پایگاه داده شامل مجموعهای از برهمکنشهای پروتئینی شناخته شده و پیش¬بینی شده براساس نتایج آزمایشگاهی، پیشبینیهای بیوانفورماتیکی و مقالات چاپ شده است. سپس مجموعه شبکه پروتئینی TNC توسط دو ابزار KEGG (https://www.genome.jp/kegg /?sess=ebfe2ad23e021e38540f798c803dd061) و Reactome (https://reactome.org/) آنالیز مسیریابی شدند. پایگاه داده Reactome مسیرهای بیولوژیکی را با جزئیات کامل نشان میدهد. همچنین KEGG بهصورت گسترده برای تجزیه و تحلیل دادهها در ژنومیک، متابولومیک و سایر مطالعات omics، مدلسازی و شبیهسازی در سیستم بیولوژی و طراحی دارو استفاده میشود. در نهایت نقش مسیرهای معنادار اصلی بهدست آمده توسط این دو روش در روند استخوانزایی ارزیابی گردید.
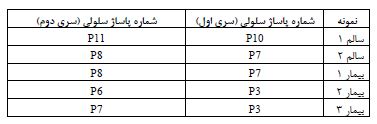
کشت و نگهداری سلولهای فیبروبلاست انسانی در شرایط طبیعی: در ابتدا بیوپسیهای پوست سه بیمار مبتلا بهاستئوژنزیزایمپرفکتا و دو فرد سالم به قطعات کوچک 2 میلیمتری بریده و توسط بافر PBS شسته شدند. سپس خراشهای عمیقی بر روی یک ظرف پتریدیش 10 سانتیمتری توسط یک تیغه استریل ایجاد شد و قطعات پوستی روی خراشهای ایجاد شده ثابت شدند. پس از آن، در محیط کشت حاوی DMEM با گلوکز بالا (HyClone,Thermo Scientific, USA, Cat. No. SH30022.01)، 10 درصد سرم گاوی جنینی (FBS) (HyClone, Thermo Scientific, USA, Cat. No. SH30070.03 )، 1 درصد پنیسیلین / استرپتومایسین (Gibco, USA, Cat. No. 15140-122) و2 میلی مولار L-گلوتامین (Thermo Fisher Scientific, USA, Cat. No. 25030081) کشت داده شدند. معمولاً 3 تا 5 روز طول میکشد تا رشد سلولهای فیبروبلاست اوّلیه از بیوپسی پوست شروع شود. مشخصات پاساژهای سلولی استفاده شده در این مطالعه در جدول 1 آمده است.
جدول 1: نمونههای مورد استفاده در این مطالعه و شماره پاساژ سلولی آنها
استخراج RNA از سلولهای فیبروبلاست کشت داده شده با استفاده از کیت RNeasy Mini (Qiagen, Germany,Cat. No 74106): سلولهای چسبیده به کف پلیت از طریق روش تریپسینه کردن جمعآوری و سلولها با استفاده از 600 میکرولیتر محلول لیزکننده RLT و با استفاده از ورتکس لیز شدند. بهمنظور همگنسازی، لیز سلولی مستقیماً بهستونهای QIAshredder spin column منتقل شده و به مدت دو دقیقه در سرعت بالا سانتریفیوژ شدند. بهاندازه حجم لیز سلولی، اتانول 70% به آن اضافه شده و از طریق پیپتاژ کردن بهطور کامل مخلوط گردید. سپس نمونه¬ها به ستون RNeasy spin column منتقل و در دور g 8000 به مدت 15 ثانیه سانتریفیوژ شدند. پس از آن بافر RW1 و RPE به ترتیب به نمونهها اضافه و در هر مرحله نمونه¬ها در دور g 8000 بهمدت 15 ثانیه سانتریفیوژ شدند. همچنین به منظور حذف تمام DNA ژنومی از روش هضم DNase برروی ستون استفاده شد. RNA استخراج شده در دمای 80- درجه سانتیگراد نگهداری گردید. سنتز cDNA با استفاده از کیتiScript reverse transcriptase (Bio-Rad, USA, Cat. No. 1708891)
حدود 1 میکروگرم RNA استخراج شده برای ساخت cDNA استفاده شد. سنتز cDNA با استفاده از کیتiScript reverse transcriptase صورت گرفت. در مرحله اوّل، RNA به لولههای PCR حاوی 4 میکرولیتر از مخلوط 5x iScript Reaction Mix و 1 میکرولیتر از iScript reverse transcriptase اضافه شد بهگونهای که مجموع حجم هر واکنش پس از اضافه کردن آب دو بار تقطیر 20 میکرولیتر گردید. پس از انکوبه کردن بهمدت 5 دقیقه در دمای اتاق، مخلوط بهدستگاه PCRمنتقل شد. در دستگاه PCR، نمونهها در دمای 46 درجه سانتیگراد بهمدت 20 دقیقه و سپس در 95 درجه سانتیگراد بهمدت 1 دقیقه انکوبه شده و در آخر نمونهها در 4 درجه سانتیگراد سرد شدند. cDNA سنتز شده در دمای 80- درجه سانتیگراد نگهداری شد و با توجه به غلظت مورد نیاز بهصورت خالص یا رقیق شده (نسبت 1 به 10) مورد استفاده قرار گرفت.
تکنیک Quantitative real-time PCR
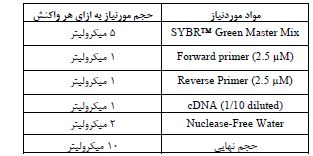
تکنیک Quantitative real-time PCR یا qRT-PCR با استفاده از سایبرگرین Applied Biosystems™ PowerUp™ SYBR™ Green Master Mix و بهصورت تکرارهای سه تایی برای هر نمونه انجام شد. در این مطالعه از ژن GAPDH به عنوان ژن خانهنگهدار برای نرمالسازی دادهها استفاده شد. تمام پرایمرها در غلظت 2.5 میکرومولار مورد استفاده قرار گرفتند. مواد تهیّه شده برای تکنیک qRT-PCR در پلیتهای 384 چاهکی (Applied Biosystems, USA, Cat. No. 4309849) ریخته و توسط فیلم چسبناکی (Thermo Scientific, UK, Cat. No. AB-0558) مهر و موم شدند. سپس تکنیکreal-time PCR در دستگاه ABI Prism 7900HT Fast Real-Time PCR System (Applied Biosystems, US) صورت گرفت. میزان مواد مورد استفاده در حجم نهایی 10 میکرولیتر و همچنین برنامه Real-time PCR به ترتیب در جدول 2 و 3 آمده است.
تعیین اختصاصیت و کارایی هر جفت پرایمر پس از انجام Real-time PCR
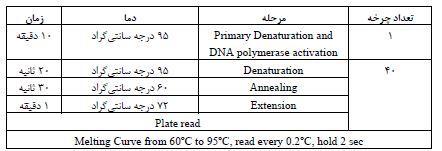
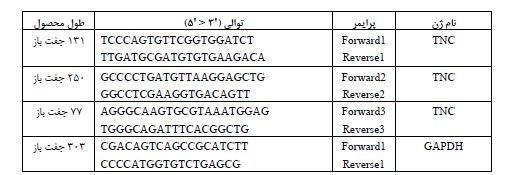
همانطور که در برنامه Real-time PCR آمده است، پس از اتمام چرخههای PCR، واکنش وارد مرحله منحنی ذوب شد. در این مرحله دما از 60 تا 95 درجه سانتیگراد بهتدریج افزایش مییابد و در دمای مخصوصی کل محصول دو رشتهای مورد نظر دناتوره شده و تک رشتهای میگردد. در صورتیکه پرایمرها اختصاصی نباشند در حین PCR، بهجز محصول اصلی، محصول یا محصولات فرعی و یا پرایمر دایمر تشکیل میگردد. وجود یک پیک در منحنی ذوب نشاندهنده اختصاصی بودن و مناسب بودن پرایمرها است. همچنین کارایی پرایمرهای طراحی شده با استفاده از منحنی استاندارد و محاسبه R2 تعیین شد. برای سنجش R2 چهار رقت متوالی از cDNA نمونه طبیعی در غلظتهای 1/8، 1/16، 1/32، 1/64 تهیه شد و سپس Real Time PCR هر رقت با هر جفت پرایمر انجام گرفت. هرچه میزان R2 به یک نزدیکتر باشد، ارتباط معنادارتری بین غلظت cDNA و Ct وجود دارد که این نشاندهنده کارایی پرایمر است. در نهایت به منظور رفع خطاهای تکنیکی، سه جفت پرایمر برای مطالعه بیان ژن TNC از طریق سایت (https://pga.mgh.harvard.edu/cgi-bin/primerbank/new_search2.cgi) PrimerBank انتخاب شدند. همچنین ژن GAPDH به عنوان ژن خانهنگه¬دار به منظور نرمالسازی دادههای Real-time PCR مورد استفاده قرار گرفت (جدول 4).
آنالیز نتایج با استفاده از روش ΔΔCT
پس از پایان واکنش و تعیین خط آستانه، سیکل آستانه (Ct) هر نمونه بهدست میآید. از نسبت سیکل آستانه ژن مورد نظر به سیکل آستانه ژن خانهنگهدار میتوان میزان بیان نسبی ژن مورد نظر را از روش 2-∆∆Ct بهدست آورد. در این روش ΔΔCt از رابطه زیر استفاده میگردد:
ΔΔCt = ΔCt sample - ΔCt control
در این رابطه ΔCt sample، اختلاف میان سیکلهای آستانه ژن مورد نظر و خانهنگهدار در نمونه مورد آزمایش (بیمار) و ΔCt control، اختلاف میان سیکلهای آستانه ژن مورد نظر و خانهنگهدار در نمونه کنترل (سالم) میباشد. عدد حاصل از آن، در رابطه 2-ΔΔCt قرار داده میشود که نتیجه بهدست آمده اختلاف میزان بیان در نمونه کنترل و مورد آزمایش را نشان میدهد.
تجزیه و تحلیل آماری
در این مطالعه دادههای جمعآوری شده مورد تجزیه و تحلیل قرار گرفتند و میزان تغییر بیان ژن TNC بین نمونههای سالم و بیمار از طریق آنالیزstudent t.test و نرمافزار Excel سنجیده شد ( p < 0/05 به عنوان سطح معناداری در نظرگرفته شده است). همچنین نمودارها توسط نرمافزارGraphPad Prism 5.0 (GraphPad Software, Inc, San Diego, CA) تهیّه شدند.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه اصفهان تایید شده است (کد اخلاق IR.UI.REC.1399.029).
جدول 2: مواد و مقادیر موردنیاز جهت انجام واکنش Real-time PCR در حجم نهایی 10 میکرولیتر
جدول 3: برنامه Real-time PCR
جدول 4: پرایمرهای طراحی شده برای Real-time PCR
نتایج
در این مطالعه مورد شاهدی، ابتدا سه بیمار خویشاوند مبتلا بهسندرم وراثتی پوکی استخوان استئوژنزیز ایمپرفکتا شناسایی شدند. شدت پوکی استخوان در افراد مبتلا بسیار بالا است بهگونهای که نوزادان در حین تولد دچار شکستگی استخوان در نواحی متعددی شدند. بیوپسی پوست از 2 فرد سالم و 3 بیمار گرفته شد و پس از کشت و جداسازی سلولهای فیبروبلاست از سلولهای کراتینوسیت، RNA سلولهای فیبروبلاست با استفاده از کیت RNeasy Mini استخراج گردید. از آنجایی که شرایط مختلف محیطی و تعداد پاساژ برروی میزان بیان ژنهای مختلف در سلول تاثیر میگذارند، در این مطالعه برای هر نمونه، RNA از سلولها در دو پاساژ مختلف استخراج شد (جدول 1). همچنین در مطالعه حاضر، برهمکنشهای پروتئینی ژن کاندید TNC در انسان توسط سایت https://string-db.org/ جمعآوری شد (شکل 1). در این برهمکنشها، پروتئینهای BMP4، ACAN، SDC4، LAMB2، NCAN، FN1، LAMC1 با پروتئین TNC در ارتباط هستند. سپس، مجموعه شبکه پروتئینی TNC توسط ابزارهای مسیریابی Reactome و KEGG، آنالیز مسیریابی شدند که مسیرهای بیولوژیکی مرتبط با استخوانزایی شامل ماتریکس خارج سلولی (ECM) Extracellular matrix و برهمکنشهای سیندکان (Sdc) Syndecan به عنوان مسیرهای معنادار اصلی شناسایی گردیدند (جدول 5). بهدلیل نقش بسیار مهم این سیگنال¬ها بهخصوص ماتریکس خارج سلولی در پاتوفیزیولوژی پوکی استخوان (12، 11) و عدم مطالعه اختصاصی ژن TNC در روند پوکی استخوان انسان، ارتباط بین بیان این ژن و پوکی استخوان در بیماران مبتلا به سندرم وراثتی پوکی استخوان استئوژنزیز ایمپرفکتا مورد بررسی قرار گرفت. بدین منظور، سه جفت پرایمر برای ژن TNC طراحی شدند که کارایی آنها با استفاده از منحنی استاندارد و محاسبه R2 تعیین گردید. پس از تهیه رقتهای مختلف، میزان R2 برای جفت پرایمر اول 0/968، برای جفت پرایمر دوم 0/64 و برای جفت پرایمر سوم 0/84 محاسبه شد. در نتیجه پرایمر اول و سوم به عنوان پرایمر کارآمدتر برای سنجش بیان ژن TNC انتخاب گردیدند. سپس، اختصاصیت هر دو جفت پرایمر با استفاده از منحنی ذوب و مشاهده یک پیک تأیید شد. در نهایت افزایش معنادار بیان ژن TNC با استفاده از نمونههای سری اول (p =0/005 ) و سری دوم (p =0/007 ) از طریق آنالیز Student t.test تأیید شد (شکل 2و 3). که میتواند نشاندهنده نقش احتمالی این ژن در روند استخوانزایی طبیعی و پوکی استخوان در دوران پیری باشد.
جدول 5: مسیرهای به دست آمده از شبکه پروتئینی TNC
شکل 1: برهمکنشهای مولکولی بین TNC و سایر پروتئینها در انسان. پروتئینهای شبکه TNC شامل ACAN، SDC4، NCAN، BMP4، FN1، IL6، ALB، C4A، LAMB2 و LAMC1 هستند که از جمله آنها میتوان به پروتئینBMP4 و ACAN اشاره کرد. پروتئینBMP4 بهصورت مستقیم در القای تشکیل استخوان و غضروف نقش دارد و همچنین پروتئوگلیکان ACAN به عنوان عضو اصلی ماتریکس خارجسلولی بافت غضروف است و در ناهنجاریهای استخوانی مثل آرتروز دخیل میباشد.

شکل 2: افزایش معنادار بیان ژن TNC در افراد بیمار نسبت به افراد سالم. در این بررسی از نمونههای سری اول و جفت پرایمر اول استفاده شده است.
p < 0/01 * اختلاف معنادار بیان TNC بین گروه افراد بیمار و سالم را نشان میدهد.
شکل 3: افزایش معنادار بیان ژن TNC در افراد بیمار نسبت به افراد سالم. در این بررسی از نمونه¬های سری دوم و جفت پرایمر سوم استفاده شده است.
p < 0/01* اختلاف معنادار بیانTNC بین گروه افراد بیمار و سالم را نشان می¬دهد.
بحث
پوکی استخوان بهعنوان یک بیماری اسکلتی سیستماتیک و اختلال مرتبط با سن تعریف میشود که با کمبود تراکم و تغییر بافت استخوانی همراه است و در نهایت منجر به افزایش شکنندگی استخوان میگردد (13). بازسازی استخوان فرایندی مداوم در طول زندگی است که در سه دهه اول، بهصورت یک ارتباط پایدار و ثابت بین جذب استخوان و تشکیل استخوان وجود دارد و با افزایش سن این تعادل به هم میخورد (14). مطالعه اختصاصی پوکی استخوان در افراد مسن بهدلیل پیچیدگی روند پیری و همراه بودن این فرایند با بیماریهای زمینهای دیگر مانند دیابت، فشار خون، قند خون و بیماریهای قلبی و عروقی بسیار دشوار است. در مقابل، بیماریهای نادر و ارثی معمولاً در اثر جهش در یک ژن ایجاد میشوند و مطالعه برروی این بیماریها سبب شناسایی مکانیسمهای اختصاصی در روند بیماریزایی میشود. در این مطالعه، سه بیمار خویشاوند مبتلا به استئوژنزیز ایمپرفکتا با پوکی استخوان بسیار شدید و شکستگیهای متعدد استخوانی شناسایی شدند و ژن TNC به عنوان کاندید اولیه انتخاب گردید. TNC یک عضو از خانواده گلیکوپروتئینهای Tenascin است. Tenascinها گلیکوپروتئینهای ماتریکس سلولی هستند که در طی تکامل و بازسازی بافت بیان میشوند. علاوهبراین، بیان Tenascinها تحت شرایط پاتولوژیک شامل زخمهای پوستی، آترواسکلروز، آسم و سرطان افزایش مییابد (5). Tenascinها همچنین تمایز و تکثیر استئوبلاست را تعدیل میکنند و در آسیبشناسیهای مربوط بهاسکلت از جمله تومورهای استخوان و استئوآرتریت نقش دارند (16، 15). این گلیکوپروتئینها نقش مهمی در چسبندگی استئوکلاست و ترمیم شکستگی دارند بهطوریکه در موش فاقد TNC در هنگام ترمیم شکستگی، جذب نامناسب و تولید بیش از حد استخوان رخ میدهد (17). در مهرهداران خانواده Tenascin دارای چهار عضو C، W، R و Xb هستند و از این میان Tenascin-C و Tenascin-W بهطور عمده در تکامل اسکلتی نقش دارند (18). در این مطالعه، ابتدا شبکه پروتئینی TNC بهدست آمد و سپس بهطور اختصاصی، این شبکه پروتئینی توسط ابزارهای KEGG و Reactome مسیریابی شدند. مسیرهای معنادار اصلی بهدست آمده بهطور کلی شامل مسیرهای مرتبط با ماتریکس خارج سلولی (ECM) و برهمکنشهای سیندکان (Sdc) هستند. ماتریکس خارج سلولی شامل طیف وسیعی از ماکرومولکولها میباشد که به انواع کلاژن، الاستین و پروتئینهای میکروفیبریلدار و همچنین پروتئوگلیکانها از جمله هیالورونان و گلیکوپروتئینهای غیرکلاژن طبقه بندی میشوند. مولکولهای ECM علاوه بر نقش ساختاری در کنترل رویدادهای مهم سلولی مانند چسبندگی، مهاجرت، تکثیر، تمایز و بقا حائز اهمیت هستند. هرگونه نقص ساختاری ارثی یا اکتسابی و یا اختلال متابولیکی در ECM ممکن است باعث ایجاد تغییرات سلولی و در نتیجه پیشرفت بیماری در بافت خاصی گردد (19). وجود بیش از صد نوع پروتئین ECM در بافت استخوان و نقش اساسی این پروتئینها در روند بازسازی آن، نشاندهنده اهمیت پروتئینهای ECM در فیزیولوژی بافت استخوان و بیماریهای وابسته به آن است. بهطور کلی، استخوانها در طول زندگی برای حفظ ساختار و عملکرد قوی بهطور مداوم بازسازی میشوند و بازسازی ناکارآمد باعث ایجاد شرایط پاتولوژیک از جمله پوکی استخوان میگردد.
پروتئینهای ECM نقش بسیار مهمی در روند بازسازی استخوان از طریق ایجاد فعالیت متعادل بین سلولهای استئوکلاست در بازجذب استخوان و سلولهای استئوبلاست در تولید استخوان جدید ایفا میکنند. همچنین، پروتئینهای ECM از جمله کلاژن نوع I میتوانند به عنوان داربستی برای اتصال مواد معدنی عمل کنند. علاوهبراین، ECM فرایند تمایز سلول¬های بنیادی مزانشیمی بهسلولهای استئوبلاست را بهطور مستقیم از طریق برهمکنش مستقیم سلول-ECM و تعدیل فعالیت فاکتور رشد کنترل میکند. بهطور مشابه، ECM میتواند بر تکامل استئوکلاستها از سلولهای پیشساز ماکروفاژ تمایز نیافته تأثیر بگذارد و عملکرد استئوکلاست را از طریق اتصال مستقیم این سلولها به اجزای ماتریکس تنظیم کند (20). نکته قابل توجه اینکه فراتر از تراکم و محتوای مواد معدنی، یکپارچگی ECM در مقاومت به شکستگی استخوان نقش دارد. پروتئینهای ECM بهطور اختصاصی برای جلوگیری از شروع و انتشار آسیبهای کوچک سازماندهی شدهاند و طی روند پیری، تغییراتی در ECM رخ میدهد که باعث شکننده شدن استخوان می¬گردد (21). علاوه بر ECM، شبکه پروتئینی TNC در مسیر مربوط به برهمکنشهای سیندکان نقش دارد. این خانواده 4 عضو دارد (Sdc 1-4) که این پروتئینهای غشایی از یک هسته پروتئینی و زنجیرههای گلیکوزآمینوگلیکان تشکیل شدهاند. هرچند این چهار سیندکان عملکردهای مشترکی دارند اما به نظر میرسد که هرکدام از آنها سیگنالهای منحصربهفرد خودشان را دارند و طبق یک الگوی اختصاصی در روند تکامل و در سلولهای مختلف بیان میشوند. به عنوان مثال، بیان پروتئین Sdc2 در طی تمایز سلول¬های استئوبلاست افزایش مییابد. از نظر مکانیسمی هم این پروتئین عملکردهای مختلفی را در سلولهای استئوبلاست به عنوان یک کمک گیرنده برای فاکتورهای رشد فیبروبلاست و پروتئینهای Wnt انجام میدهد و چسبندگی سلولی، تکثیر، تمایز و آپوپتوز را کنترل میکند. مطالعات اخیر همچنین نشان میدهد که پروتئین Sdc2 از طریق تعامل با سیگنال Wnt / β-catenin به پاسخ سلولهای استئوسارکوما به عوامل سمی سلولی کمک میکند. علاوهبراین، نقش پروتئین Sdc2 در پاتولوژی استئوسارکوما و سایر تومورها گزارش شده است (22) و بررسیها نشان میدهد که Sdc2 از جمله ژنهایی هست که سهم بسیار مهمی در طی روند بهبودی شکستگی در پوکی استخوان ایفا میکند (23). همچنین یافتههای یک گروه از دانشمندان نشاندهنده نقش حیاتی پروتئین Sdc3 در متابولیسم استخوان از طریق افزایش سیگنال Wnt در سلولهای استئوبلاست و استئوکلاست است. نتایج آن¬ها پروتئین Sdc3 را به عنوان یک هدف درمانی جدید برای توسعه داروی جدید آنابولیک برای درمان پوکی استخوان معرفی میکند (24). علاوهبراین، پروتئین Sdc4 که بهطور مستقیم در شبکه پروتئینی TNC قرار گرفته با بیماری استخوانی آرتروز در ارتباط است و از طریق دخیل بودن در استخوان¬زایی اندوکندرال در فرایند ترمیم شکستگی نقش دارد (25). به دلیل ارتباط مستقیم سیگنالهای ECM و سیندکان با روند استخوانزایی و پوکی استخوان، ژن TNC به عنوان کاندید نهایی این مطالعه انتخاب گردید. سپس، برای بررسی ارتباط این ژن با پوکی استخوان، نمونه پوست از افراد مبتلا بهپوکی استخوان وراثتی استئوژنزیز ایمپرفکتا و افراد سالم گرفته شد. پس از جداسازی سلولهای فیبروبلاست نمونهها و استخراج RNA، افزایش معنادار بیان ژن TNC در افراد بیمار توسط Real-time PCR و در تکرارهای مختلف بیولوژیکی و تکنیکی تأیید شد. تاکنون مطالعات گوناگونی برروی بیان ژن TNC در بیماری¬های مختلف استخوانی صورت گرفته است که نشاندهنده افزایش میزان این پروتئین در اختلالات متعدد استخوانی هستند. Ozkan و همکارانش در سال 2015 افزایش معنادار بیان TNC را برروی بیماران مبتلا به بیماری مزمن کلیوی همراه با نقص استخوانی (CKD-MBD) Chronic kidney disease-mineral and bone disorder نشان دادند و به این نتیجه رسیدند که می¬توان این ژن را به عنوان یک بیومارکر برای تشخیص این بیماری در نظر گرفت (26). همچنین Page و همکارانش در سال 2012 گزارش کردند که می¬توان TNC را به عنوان بیومارکر فرسایش استخوانی معرفی کرد زیرا سطح پروتئین TNC در افراد مبتلا به روماتیسم مفصلی بهطور قابل ملاحظهای بالاست و بالاترین میزان آن در مرحله نهایی روماتیسم مشاهده شد (27). علاوهبراین، بر اساس مطالعات انجام شده توسط Patel و همکارانش در سال 2011 میزان TNC در مایع سینوویال و غضروف زانو بیماران مبتلا به آرتروز بهطور قابل ملاحظهای افزایش یافته است. یافته¬های آن¬ها نشان می¬دهد که سطح بالای TNC میتواند واسطههای التهابی را القا کند و باعث تخریب ماتریکس در مفاصل بیماران مبتلا بهآرتروز شود (28). براساس اطلاعات بهدست آمده تاکنون هیچ مطالعهای برروی ارتباط TNC با بیماری پوکی استخوان در انسان صورت نگرفته است و تنها مطالعه برروی نقش TNC در پوکی استخوان موش توسط Chen و همکارانش در سال 2016 انجام گرفت و یافتههای آنها بر خلاف نتایج به دست آمده در این تحقیق نشان داد که کاهش بیان این پروتئین باعث تشدید پوکی استخوان با مهار سیگنال Wnt و سرکوب استخوانسازی میشود (29). این تضاد میتواند پیشنهاددهنده پیچیدگی بیان TNC در انواع مختلف پوکی استخوان و یا عدم کارآمدی مدل آزمایشگاهی موش برای مطالعه پوکی استخوان در انسان باشد. همچنین سنجش بیان ژن TNC برروی بیماران بیشتر مبتلا به انواع پوکی استخوان برای تأیید دادههای این مطالعه و معرفی نهایی این ژن به عنوان یک بیومارکر پوکی استخوان لازم است و در صورت تأیید افزایش میزان TNC در این بیماران، مهار بیان این ژن میتواند به عنوان یک روش کاندید برای درمان پوکی استخوان مورد مطالعه قرار گیرد.
نتیجهگیری
در این مطالعه، ارتباط بین ژن TNC و پوکی استخوان در بیماران مبتلا به سندرم پوکی استخوان بسیار نادر و وراثتی استئوژنزیز ایمپرفکتا بررسی گردید و افزایش معنادار بیان ژن TNC در بیماران نسبت به افراد سالم مشاهده شد. این تغییر بیان میتواند نشاندهنده نقش احتمالی ژن TNC در روند استخوانزایی و پوکی استخوان طبیعی در دوران پیری باشد و این ژن را به عنوان یک بیومارکر تشخیصی معرفی کند.
سپاسگزاری
این مقاله برگرفته از پایاننامه دانشگاه اصفهان میباشد. نویسندگان تحقیق حاضر از دکتر محمد شبول به دلیل شناسایی بیماران و تهیه نمونههای انسانی از کشور اردن کمال تشکر را دارند.
حامی مالی: دانشگاه اصفهان
تعارض در منافع: وجود ندارد.
پوکی استخوان بهعنوان یکی از بیماریهای مهم قرن بیست و یکم در نظر گرفته میشود و تقریباً 200 میلیون نفر در سطح جهان به این بیماری مبتلا هستند که تأثیر قابل توجهی در میزان مرگ و میر دارد (1). عمدتاً پوکی استخوان با کاهش تراکم مواد معدنی استخوان (BMD) Bone mineral density و افزایش خطر شکنندگی و وخیمتر شدن وضعیت استخوان همراه است و متأسفانه این اپیدمی ویرانگر بهطور خاموش پیش میرود تا زمانی که یک شکستگی رخ دهد (2). با توجه بهارتباط نزدیک بین روند پیری و پاتوژنز پوکی استخوان، تحقیقات در مورد مکانیسمهای از بین رفتن استخوان همراه با افزایش سن در سالهای اخیر بهطور قابل توجهی گسترش یافته است که شامل ترکیبی از مطالعات بالینی و مولکولی است. بهطور کلی، پیری یک فرایند پیچیده است که در اثر مجموعهای از فاکتورهای محیطی و ژنتیکی روی میدهد. در روند پیری، مجموعهای از تغییرات زیانآور در سلولها و بافتها انباشته میشوند که در نهایت منجر به افزایش خطر بیماری و مرگ میگردند (3). در مقابل پیری و بیماریهای رایج مثل دیابت، چاقی و بسیاری از انواع سرطانها که معمولاً در اثر مجموعهای از علائم متعدد ژنتیکی و فاکتورهای محیطی ایجاد میشوند، بیماریهای نادر وجود دارند که معمولاً در اثر جهش در یک ژن ایجاد میشوند و اثبات روند بیماریزایی آن سادهتر است. کشف مکانیسمهای دخیل در بیماریهای نادر میتواند به فهم عمیقتر ما از بیماریهای رایج مرتبط و مکانیسمهای مولکولی آنها کمک کند. برای مثال گرچه بیماران مبتلا به بیماری نادر و وراثتی پوکی استخوان درصد بسیار کمی از جمعیّت را تشکیل میدهند، امّا فرایند پوکی استخوان با افزایش سن بهصورت رایج در زندگی بسیاری از انسانها وجود دارد و تلاشهای بسیار زیادی تاکنون برای پیشگیری و درمان آن صورت گرفته است. مطالعه بر روی بیماری نادر پوکی استخوان یک ابزار بسیار مفید برای مطالعه اختصاصی روند پوکی استخوان در دوران پیری است بدین صورت که ژنهای دخیل در بیماری و مکانیسم آنها میتوانند در طراحی بسیاری از درمانها مورد استفاده قرار گیرند. از جمله بیماریهای ارثی و مونوژنیک شناخته شده در ارتباط با پوکی استخوان میتوان به استئوژنزیز ایمپرفکتا (osteogenesis imperfecta)، سندرم بروک (Bruck syndrome)، استئوپتروزیس (osteopetrosis)، سندرم استئوپورزیس-سودوگلیوما (osteoporosis-pseudoglioma syndrome)، بیماری ون بوخم (von Buchem disease)، اسکلروستئوزتئوزیز (sclerosteosteosis)، استئولیز خانوادگی (familial osteolysis)، بیماری پاژه (Paget’s disease)، هیپوفسفاتازی (hypophosphatasia)، هایپرپاراتیروئیدیسم نوزادی(neonatal hyperparathyroidism) و پیکنودیزوستوز (pyknodysostosis) اشاره کرد (4). در این مطالعه از نمونههای استئوژنزیز ایمپرفکتا استفاده گردید که در آن بیماران از بدو تولد از پوکی استخوان شدید و شکستگیهای متعدد استخوانی رنج می¬برند.
همچنین ژن (TNC) Tenascin C به عنوان ژن کاندید در روند پوکیاستخوان انتخاب شد. ژنTNC یک گلیکوپروتئین ماتریکس خارج سلولی را کد میکند که در طی رشد استخوان و مورفوژنز توسط سلولهای استئوبلاست ساخته میشود (6،5). بررسیها نشان داده است که TNC در تمایز استخوانی استئوبلاست و معدنی شدن بافت استخوان نقش دارد. در طی این تمایز، ممکن است TNC به عنوان یک واسطه برای تشکیل استخوان جدید توسط سیگنال TGF-β فعال شود (8، 7). علاوه بر این، پروتئین (BMP) Bone morphogenetic protein و فاکتورهای رشد Wnt یا فشار مکانیکی و استرس میتوانند بیان TNC را در استئوبلاستها افزایش دهند (10، 9). هدف اصلی این مطالعه بررسی ارتباط ژن TNC با بیماری پوکی استخوان با استفاده از روشهای بیوانفورماتیکی و آزمایشگاهی است. لازم بهذکر است که این مطالعه به منظور کاندید کردن بیومارکر پوکی استخوان صورت گرفته است و بهدلیل تعداد بسیار محدود نمونههای بیماران مبتلا بهاستئوژنزیز ایمپرفکتا به عنوان مدل مونوژن و نادر پوکی استخوان، لازم است این نتایج در مطالعات گستردهتر با جامعه آماری مناسب متشکل از بیماران مبتلا به انواع پوکی استخوان و افراد سالم تأیید نهایی شود.
روش بررسی
مطالعات بیوانفورماتیک و زیست شناسی سامانهها
به منظور به دست آوردن برهمکنشهای پروتئینی TNC از پایگاه داده زیستی String (https://string-db.org/) استفاده گردید. این پایگاه داده شامل مجموعهای از برهمکنشهای پروتئینی شناخته شده و پیش¬بینی شده براساس نتایج آزمایشگاهی، پیشبینیهای بیوانفورماتیکی و مقالات چاپ شده است. سپس مجموعه شبکه پروتئینی TNC توسط دو ابزار KEGG (https://www.genome.jp/kegg /?sess=ebfe2ad23e021e38540f798c803dd061) و Reactome (https://reactome.org/) آنالیز مسیریابی شدند. پایگاه داده Reactome مسیرهای بیولوژیکی را با جزئیات کامل نشان میدهد. همچنین KEGG بهصورت گسترده برای تجزیه و تحلیل دادهها در ژنومیک، متابولومیک و سایر مطالعات omics، مدلسازی و شبیهسازی در سیستم بیولوژی و طراحی دارو استفاده میشود. در نهایت نقش مسیرهای معنادار اصلی بهدست آمده توسط این دو روش در روند استخوانزایی ارزیابی گردید.
کشت و نگهداری سلولهای فیبروبلاست انسانی در شرایط طبیعی: در ابتدا بیوپسیهای پوست سه بیمار مبتلا بهاستئوژنزیزایمپرفکتا و دو فرد سالم به قطعات کوچک 2 میلیمتری بریده و توسط بافر PBS شسته شدند. سپس خراشهای عمیقی بر روی یک ظرف پتریدیش 10 سانتیمتری توسط یک تیغه استریل ایجاد شد و قطعات پوستی روی خراشهای ایجاد شده ثابت شدند. پس از آن، در محیط کشت حاوی DMEM با گلوکز بالا (HyClone,Thermo Scientific, USA, Cat. No. SH30022.01)، 10 درصد سرم گاوی جنینی (FBS) (HyClone, Thermo Scientific, USA, Cat. No. SH30070.03 )، 1 درصد پنیسیلین / استرپتومایسین (Gibco, USA, Cat. No. 15140-122) و2 میلی مولار L-گلوتامین (Thermo Fisher Scientific, USA, Cat. No. 25030081) کشت داده شدند. معمولاً 3 تا 5 روز طول میکشد تا رشد سلولهای فیبروبلاست اوّلیه از بیوپسی پوست شروع شود. مشخصات پاساژهای سلولی استفاده شده در این مطالعه در جدول 1 آمده است.
جدول 1: نمونههای مورد استفاده در این مطالعه و شماره پاساژ سلولی آنها

استخراج RNA از سلولهای فیبروبلاست کشت داده شده با استفاده از کیت RNeasy Mini (Qiagen, Germany,Cat. No 74106): سلولهای چسبیده به کف پلیت از طریق روش تریپسینه کردن جمعآوری و سلولها با استفاده از 600 میکرولیتر محلول لیزکننده RLT و با استفاده از ورتکس لیز شدند. بهمنظور همگنسازی، لیز سلولی مستقیماً بهستونهای QIAshredder spin column منتقل شده و به مدت دو دقیقه در سرعت بالا سانتریفیوژ شدند. بهاندازه حجم لیز سلولی، اتانول 70% به آن اضافه شده و از طریق پیپتاژ کردن بهطور کامل مخلوط گردید. سپس نمونه¬ها به ستون RNeasy spin column منتقل و در دور g 8000 به مدت 15 ثانیه سانتریفیوژ شدند. پس از آن بافر RW1 و RPE به ترتیب به نمونهها اضافه و در هر مرحله نمونه¬ها در دور g 8000 بهمدت 15 ثانیه سانتریفیوژ شدند. همچنین به منظور حذف تمام DNA ژنومی از روش هضم DNase برروی ستون استفاده شد. RNA استخراج شده در دمای 80- درجه سانتیگراد نگهداری گردید. سنتز cDNA با استفاده از کیتiScript reverse transcriptase (Bio-Rad, USA, Cat. No. 1708891)
حدود 1 میکروگرم RNA استخراج شده برای ساخت cDNA استفاده شد. سنتز cDNA با استفاده از کیتiScript reverse transcriptase صورت گرفت. در مرحله اوّل، RNA به لولههای PCR حاوی 4 میکرولیتر از مخلوط 5x iScript Reaction Mix و 1 میکرولیتر از iScript reverse transcriptase اضافه شد بهگونهای که مجموع حجم هر واکنش پس از اضافه کردن آب دو بار تقطیر 20 میکرولیتر گردید. پس از انکوبه کردن بهمدت 5 دقیقه در دمای اتاق، مخلوط بهدستگاه PCRمنتقل شد. در دستگاه PCR، نمونهها در دمای 46 درجه سانتیگراد بهمدت 20 دقیقه و سپس در 95 درجه سانتیگراد بهمدت 1 دقیقه انکوبه شده و در آخر نمونهها در 4 درجه سانتیگراد سرد شدند. cDNA سنتز شده در دمای 80- درجه سانتیگراد نگهداری شد و با توجه به غلظت مورد نیاز بهصورت خالص یا رقیق شده (نسبت 1 به 10) مورد استفاده قرار گرفت.
تکنیک Quantitative real-time PCR
تکنیک Quantitative real-time PCR یا qRT-PCR با استفاده از سایبرگرین Applied Biosystems™ PowerUp™ SYBR™ Green Master Mix و بهصورت تکرارهای سه تایی برای هر نمونه انجام شد. در این مطالعه از ژن GAPDH به عنوان ژن خانهنگهدار برای نرمالسازی دادهها استفاده شد. تمام پرایمرها در غلظت 2.5 میکرومولار مورد استفاده قرار گرفتند. مواد تهیّه شده برای تکنیک qRT-PCR در پلیتهای 384 چاهکی (Applied Biosystems, USA, Cat. No. 4309849) ریخته و توسط فیلم چسبناکی (Thermo Scientific, UK, Cat. No. AB-0558) مهر و موم شدند. سپس تکنیکreal-time PCR در دستگاه ABI Prism 7900HT Fast Real-Time PCR System (Applied Biosystems, US) صورت گرفت. میزان مواد مورد استفاده در حجم نهایی 10 میکرولیتر و همچنین برنامه Real-time PCR به ترتیب در جدول 2 و 3 آمده است.
تعیین اختصاصیت و کارایی هر جفت پرایمر پس از انجام Real-time PCR
همانطور که در برنامه Real-time PCR آمده است، پس از اتمام چرخههای PCR، واکنش وارد مرحله منحنی ذوب شد. در این مرحله دما از 60 تا 95 درجه سانتیگراد بهتدریج افزایش مییابد و در دمای مخصوصی کل محصول دو رشتهای مورد نظر دناتوره شده و تک رشتهای میگردد. در صورتیکه پرایمرها اختصاصی نباشند در حین PCR، بهجز محصول اصلی، محصول یا محصولات فرعی و یا پرایمر دایمر تشکیل میگردد. وجود یک پیک در منحنی ذوب نشاندهنده اختصاصی بودن و مناسب بودن پرایمرها است. همچنین کارایی پرایمرهای طراحی شده با استفاده از منحنی استاندارد و محاسبه R2 تعیین شد. برای سنجش R2 چهار رقت متوالی از cDNA نمونه طبیعی در غلظتهای 1/8، 1/16، 1/32، 1/64 تهیه شد و سپس Real Time PCR هر رقت با هر جفت پرایمر انجام گرفت. هرچه میزان R2 به یک نزدیکتر باشد، ارتباط معنادارتری بین غلظت cDNA و Ct وجود دارد که این نشاندهنده کارایی پرایمر است. در نهایت به منظور رفع خطاهای تکنیکی، سه جفت پرایمر برای مطالعه بیان ژن TNC از طریق سایت (https://pga.mgh.harvard.edu/cgi-bin/primerbank/new_search2.cgi) PrimerBank انتخاب شدند. همچنین ژن GAPDH به عنوان ژن خانهنگه¬دار به منظور نرمالسازی دادههای Real-time PCR مورد استفاده قرار گرفت (جدول 4).
آنالیز نتایج با استفاده از روش ΔΔCT
پس از پایان واکنش و تعیین خط آستانه، سیکل آستانه (Ct) هر نمونه بهدست میآید. از نسبت سیکل آستانه ژن مورد نظر به سیکل آستانه ژن خانهنگهدار میتوان میزان بیان نسبی ژن مورد نظر را از روش 2-∆∆Ct بهدست آورد. در این روش ΔΔCt از رابطه زیر استفاده میگردد:
ΔΔCt = ΔCt sample - ΔCt control
در این رابطه ΔCt sample، اختلاف میان سیکلهای آستانه ژن مورد نظر و خانهنگهدار در نمونه مورد آزمایش (بیمار) و ΔCt control، اختلاف میان سیکلهای آستانه ژن مورد نظر و خانهنگهدار در نمونه کنترل (سالم) میباشد. عدد حاصل از آن، در رابطه 2-ΔΔCt قرار داده میشود که نتیجه بهدست آمده اختلاف میزان بیان در نمونه کنترل و مورد آزمایش را نشان میدهد.
تجزیه و تحلیل آماری
در این مطالعه دادههای جمعآوری شده مورد تجزیه و تحلیل قرار گرفتند و میزان تغییر بیان ژن TNC بین نمونههای سالم و بیمار از طریق آنالیزstudent t.test و نرمافزار Excel سنجیده شد ( p < 0/05 به عنوان سطح معناداری در نظرگرفته شده است). همچنین نمودارها توسط نرمافزارGraphPad Prism 5.0 (GraphPad Software, Inc, San Diego, CA) تهیّه شدند.
ملاحظات اخلاقی
پروپوزال این تحقیق توسط دانشگاه اصفهان تایید شده است (کد اخلاق IR.UI.REC.1399.029).
جدول 2: مواد و مقادیر موردنیاز جهت انجام واکنش Real-time PCR در حجم نهایی 10 میکرولیتر

جدول 3: برنامه Real-time PCR

جدول 4: پرایمرهای طراحی شده برای Real-time PCR

نتایج
در این مطالعه مورد شاهدی، ابتدا سه بیمار خویشاوند مبتلا بهسندرم وراثتی پوکی استخوان استئوژنزیز ایمپرفکتا شناسایی شدند. شدت پوکی استخوان در افراد مبتلا بسیار بالا است بهگونهای که نوزادان در حین تولد دچار شکستگی استخوان در نواحی متعددی شدند. بیوپسی پوست از 2 فرد سالم و 3 بیمار گرفته شد و پس از کشت و جداسازی سلولهای فیبروبلاست از سلولهای کراتینوسیت، RNA سلولهای فیبروبلاست با استفاده از کیت RNeasy Mini استخراج گردید. از آنجایی که شرایط مختلف محیطی و تعداد پاساژ برروی میزان بیان ژنهای مختلف در سلول تاثیر میگذارند، در این مطالعه برای هر نمونه، RNA از سلولها در دو پاساژ مختلف استخراج شد (جدول 1). همچنین در مطالعه حاضر، برهمکنشهای پروتئینی ژن کاندید TNC در انسان توسط سایت https://string-db.org/ جمعآوری شد (شکل 1). در این برهمکنشها، پروتئینهای BMP4، ACAN، SDC4، LAMB2، NCAN، FN1، LAMC1 با پروتئین TNC در ارتباط هستند. سپس، مجموعه شبکه پروتئینی TNC توسط ابزارهای مسیریابی Reactome و KEGG، آنالیز مسیریابی شدند که مسیرهای بیولوژیکی مرتبط با استخوانزایی شامل ماتریکس خارج سلولی (ECM) Extracellular matrix و برهمکنشهای سیندکان (Sdc) Syndecan به عنوان مسیرهای معنادار اصلی شناسایی گردیدند (جدول 5). بهدلیل نقش بسیار مهم این سیگنال¬ها بهخصوص ماتریکس خارج سلولی در پاتوفیزیولوژی پوکی استخوان (12، 11) و عدم مطالعه اختصاصی ژن TNC در روند پوکی استخوان انسان، ارتباط بین بیان این ژن و پوکی استخوان در بیماران مبتلا به سندرم وراثتی پوکی استخوان استئوژنزیز ایمپرفکتا مورد بررسی قرار گرفت. بدین منظور، سه جفت پرایمر برای ژن TNC طراحی شدند که کارایی آنها با استفاده از منحنی استاندارد و محاسبه R2 تعیین گردید. پس از تهیه رقتهای مختلف، میزان R2 برای جفت پرایمر اول 0/968، برای جفت پرایمر دوم 0/64 و برای جفت پرایمر سوم 0/84 محاسبه شد. در نتیجه پرایمر اول و سوم به عنوان پرایمر کارآمدتر برای سنجش بیان ژن TNC انتخاب گردیدند. سپس، اختصاصیت هر دو جفت پرایمر با استفاده از منحنی ذوب و مشاهده یک پیک تأیید شد. در نهایت افزایش معنادار بیان ژن TNC با استفاده از نمونههای سری اول (p =0/005 ) و سری دوم (p =0/007 ) از طریق آنالیز Student t.test تأیید شد (شکل 2و 3). که میتواند نشاندهنده نقش احتمالی این ژن در روند استخوانزایی طبیعی و پوکی استخوان در دوران پیری باشد.
جدول 5: مسیرهای به دست آمده از شبکه پروتئینی TNC


شکل 1: برهمکنشهای مولکولی بین TNC و سایر پروتئینها در انسان. پروتئینهای شبکه TNC شامل ACAN، SDC4، NCAN، BMP4، FN1، IL6، ALB، C4A، LAMB2 و LAMC1 هستند که از جمله آنها میتوان به پروتئینBMP4 و ACAN اشاره کرد. پروتئینBMP4 بهصورت مستقیم در القای تشکیل استخوان و غضروف نقش دارد و همچنین پروتئوگلیکان ACAN به عنوان عضو اصلی ماتریکس خارجسلولی بافت غضروف است و در ناهنجاریهای استخوانی مثل آرتروز دخیل میباشد.

شکل 2: افزایش معنادار بیان ژن TNC در افراد بیمار نسبت به افراد سالم. در این بررسی از نمونههای سری اول و جفت پرایمر اول استفاده شده است.
p < 0/01 * اختلاف معنادار بیان TNC بین گروه افراد بیمار و سالم را نشان میدهد.

شکل 3: افزایش معنادار بیان ژن TNC در افراد بیمار نسبت به افراد سالم. در این بررسی از نمونه¬های سری دوم و جفت پرایمر سوم استفاده شده است.
p < 0/01* اختلاف معنادار بیانTNC بین گروه افراد بیمار و سالم را نشان می¬دهد.
بحث
پوکی استخوان بهعنوان یک بیماری اسکلتی سیستماتیک و اختلال مرتبط با سن تعریف میشود که با کمبود تراکم و تغییر بافت استخوانی همراه است و در نهایت منجر به افزایش شکنندگی استخوان میگردد (13). بازسازی استخوان فرایندی مداوم در طول زندگی است که در سه دهه اول، بهصورت یک ارتباط پایدار و ثابت بین جذب استخوان و تشکیل استخوان وجود دارد و با افزایش سن این تعادل به هم میخورد (14). مطالعه اختصاصی پوکی استخوان در افراد مسن بهدلیل پیچیدگی روند پیری و همراه بودن این فرایند با بیماریهای زمینهای دیگر مانند دیابت، فشار خون، قند خون و بیماریهای قلبی و عروقی بسیار دشوار است. در مقابل، بیماریهای نادر و ارثی معمولاً در اثر جهش در یک ژن ایجاد میشوند و مطالعه برروی این بیماریها سبب شناسایی مکانیسمهای اختصاصی در روند بیماریزایی میشود. در این مطالعه، سه بیمار خویشاوند مبتلا به استئوژنزیز ایمپرفکتا با پوکی استخوان بسیار شدید و شکستگیهای متعدد استخوانی شناسایی شدند و ژن TNC به عنوان کاندید اولیه انتخاب گردید. TNC یک عضو از خانواده گلیکوپروتئینهای Tenascin است. Tenascinها گلیکوپروتئینهای ماتریکس سلولی هستند که در طی تکامل و بازسازی بافت بیان میشوند. علاوهبراین، بیان Tenascinها تحت شرایط پاتولوژیک شامل زخمهای پوستی، آترواسکلروز، آسم و سرطان افزایش مییابد (5). Tenascinها همچنین تمایز و تکثیر استئوبلاست را تعدیل میکنند و در آسیبشناسیهای مربوط بهاسکلت از جمله تومورهای استخوان و استئوآرتریت نقش دارند (16، 15). این گلیکوپروتئینها نقش مهمی در چسبندگی استئوکلاست و ترمیم شکستگی دارند بهطوریکه در موش فاقد TNC در هنگام ترمیم شکستگی، جذب نامناسب و تولید بیش از حد استخوان رخ میدهد (17). در مهرهداران خانواده Tenascin دارای چهار عضو C، W، R و Xb هستند و از این میان Tenascin-C و Tenascin-W بهطور عمده در تکامل اسکلتی نقش دارند (18). در این مطالعه، ابتدا شبکه پروتئینی TNC بهدست آمد و سپس بهطور اختصاصی، این شبکه پروتئینی توسط ابزارهای KEGG و Reactome مسیریابی شدند. مسیرهای معنادار اصلی بهدست آمده بهطور کلی شامل مسیرهای مرتبط با ماتریکس خارج سلولی (ECM) و برهمکنشهای سیندکان (Sdc) هستند. ماتریکس خارج سلولی شامل طیف وسیعی از ماکرومولکولها میباشد که به انواع کلاژن، الاستین و پروتئینهای میکروفیبریلدار و همچنین پروتئوگلیکانها از جمله هیالورونان و گلیکوپروتئینهای غیرکلاژن طبقه بندی میشوند. مولکولهای ECM علاوه بر نقش ساختاری در کنترل رویدادهای مهم سلولی مانند چسبندگی، مهاجرت، تکثیر، تمایز و بقا حائز اهمیت هستند. هرگونه نقص ساختاری ارثی یا اکتسابی و یا اختلال متابولیکی در ECM ممکن است باعث ایجاد تغییرات سلولی و در نتیجه پیشرفت بیماری در بافت خاصی گردد (19). وجود بیش از صد نوع پروتئین ECM در بافت استخوان و نقش اساسی این پروتئینها در روند بازسازی آن، نشاندهنده اهمیت پروتئینهای ECM در فیزیولوژی بافت استخوان و بیماریهای وابسته به آن است. بهطور کلی، استخوانها در طول زندگی برای حفظ ساختار و عملکرد قوی بهطور مداوم بازسازی میشوند و بازسازی ناکارآمد باعث ایجاد شرایط پاتولوژیک از جمله پوکی استخوان میگردد.
پروتئینهای ECM نقش بسیار مهمی در روند بازسازی استخوان از طریق ایجاد فعالیت متعادل بین سلولهای استئوکلاست در بازجذب استخوان و سلولهای استئوبلاست در تولید استخوان جدید ایفا میکنند. همچنین، پروتئینهای ECM از جمله کلاژن نوع I میتوانند به عنوان داربستی برای اتصال مواد معدنی عمل کنند. علاوهبراین، ECM فرایند تمایز سلول¬های بنیادی مزانشیمی بهسلولهای استئوبلاست را بهطور مستقیم از طریق برهمکنش مستقیم سلول-ECM و تعدیل فعالیت فاکتور رشد کنترل میکند. بهطور مشابه، ECM میتواند بر تکامل استئوکلاستها از سلولهای پیشساز ماکروفاژ تمایز نیافته تأثیر بگذارد و عملکرد استئوکلاست را از طریق اتصال مستقیم این سلولها به اجزای ماتریکس تنظیم کند (20). نکته قابل توجه اینکه فراتر از تراکم و محتوای مواد معدنی، یکپارچگی ECM در مقاومت به شکستگی استخوان نقش دارد. پروتئینهای ECM بهطور اختصاصی برای جلوگیری از شروع و انتشار آسیبهای کوچک سازماندهی شدهاند و طی روند پیری، تغییراتی در ECM رخ میدهد که باعث شکننده شدن استخوان می¬گردد (21). علاوه بر ECM، شبکه پروتئینی TNC در مسیر مربوط به برهمکنشهای سیندکان نقش دارد. این خانواده 4 عضو دارد (Sdc 1-4) که این پروتئینهای غشایی از یک هسته پروتئینی و زنجیرههای گلیکوزآمینوگلیکان تشکیل شدهاند. هرچند این چهار سیندکان عملکردهای مشترکی دارند اما به نظر میرسد که هرکدام از آنها سیگنالهای منحصربهفرد خودشان را دارند و طبق یک الگوی اختصاصی در روند تکامل و در سلولهای مختلف بیان میشوند. به عنوان مثال، بیان پروتئین Sdc2 در طی تمایز سلول¬های استئوبلاست افزایش مییابد. از نظر مکانیسمی هم این پروتئین عملکردهای مختلفی را در سلولهای استئوبلاست به عنوان یک کمک گیرنده برای فاکتورهای رشد فیبروبلاست و پروتئینهای Wnt انجام میدهد و چسبندگی سلولی، تکثیر، تمایز و آپوپتوز را کنترل میکند. مطالعات اخیر همچنین نشان میدهد که پروتئین Sdc2 از طریق تعامل با سیگنال Wnt / β-catenin به پاسخ سلولهای استئوسارکوما به عوامل سمی سلولی کمک میکند. علاوهبراین، نقش پروتئین Sdc2 در پاتولوژی استئوسارکوما و سایر تومورها گزارش شده است (22) و بررسیها نشان میدهد که Sdc2 از جمله ژنهایی هست که سهم بسیار مهمی در طی روند بهبودی شکستگی در پوکی استخوان ایفا میکند (23). همچنین یافتههای یک گروه از دانشمندان نشاندهنده نقش حیاتی پروتئین Sdc3 در متابولیسم استخوان از طریق افزایش سیگنال Wnt در سلولهای استئوبلاست و استئوکلاست است. نتایج آن¬ها پروتئین Sdc3 را به عنوان یک هدف درمانی جدید برای توسعه داروی جدید آنابولیک برای درمان پوکی استخوان معرفی میکند (24). علاوهبراین، پروتئین Sdc4 که بهطور مستقیم در شبکه پروتئینی TNC قرار گرفته با بیماری استخوانی آرتروز در ارتباط است و از طریق دخیل بودن در استخوان¬زایی اندوکندرال در فرایند ترمیم شکستگی نقش دارد (25). به دلیل ارتباط مستقیم سیگنالهای ECM و سیندکان با روند استخوانزایی و پوکی استخوان، ژن TNC به عنوان کاندید نهایی این مطالعه انتخاب گردید. سپس، برای بررسی ارتباط این ژن با پوکی استخوان، نمونه پوست از افراد مبتلا بهپوکی استخوان وراثتی استئوژنزیز ایمپرفکتا و افراد سالم گرفته شد. پس از جداسازی سلولهای فیبروبلاست نمونهها و استخراج RNA، افزایش معنادار بیان ژن TNC در افراد بیمار توسط Real-time PCR و در تکرارهای مختلف بیولوژیکی و تکنیکی تأیید شد. تاکنون مطالعات گوناگونی برروی بیان ژن TNC در بیماری¬های مختلف استخوانی صورت گرفته است که نشاندهنده افزایش میزان این پروتئین در اختلالات متعدد استخوانی هستند. Ozkan و همکارانش در سال 2015 افزایش معنادار بیان TNC را برروی بیماران مبتلا به بیماری مزمن کلیوی همراه با نقص استخوانی (CKD-MBD) Chronic kidney disease-mineral and bone disorder نشان دادند و به این نتیجه رسیدند که می¬توان این ژن را به عنوان یک بیومارکر برای تشخیص این بیماری در نظر گرفت (26). همچنین Page و همکارانش در سال 2012 گزارش کردند که می¬توان TNC را به عنوان بیومارکر فرسایش استخوانی معرفی کرد زیرا سطح پروتئین TNC در افراد مبتلا به روماتیسم مفصلی بهطور قابل ملاحظهای بالاست و بالاترین میزان آن در مرحله نهایی روماتیسم مشاهده شد (27). علاوهبراین، بر اساس مطالعات انجام شده توسط Patel و همکارانش در سال 2011 میزان TNC در مایع سینوویال و غضروف زانو بیماران مبتلا به آرتروز بهطور قابل ملاحظهای افزایش یافته است. یافته¬های آن¬ها نشان می¬دهد که سطح بالای TNC میتواند واسطههای التهابی را القا کند و باعث تخریب ماتریکس در مفاصل بیماران مبتلا بهآرتروز شود (28). براساس اطلاعات بهدست آمده تاکنون هیچ مطالعهای برروی ارتباط TNC با بیماری پوکی استخوان در انسان صورت نگرفته است و تنها مطالعه برروی نقش TNC در پوکی استخوان موش توسط Chen و همکارانش در سال 2016 انجام گرفت و یافتههای آنها بر خلاف نتایج به دست آمده در این تحقیق نشان داد که کاهش بیان این پروتئین باعث تشدید پوکی استخوان با مهار سیگنال Wnt و سرکوب استخوانسازی میشود (29). این تضاد میتواند پیشنهاددهنده پیچیدگی بیان TNC در انواع مختلف پوکی استخوان و یا عدم کارآمدی مدل آزمایشگاهی موش برای مطالعه پوکی استخوان در انسان باشد. همچنین سنجش بیان ژن TNC برروی بیماران بیشتر مبتلا به انواع پوکی استخوان برای تأیید دادههای این مطالعه و معرفی نهایی این ژن به عنوان یک بیومارکر پوکی استخوان لازم است و در صورت تأیید افزایش میزان TNC در این بیماران، مهار بیان این ژن میتواند به عنوان یک روش کاندید برای درمان پوکی استخوان مورد مطالعه قرار گیرد.
نتیجهگیری
در این مطالعه، ارتباط بین ژن TNC و پوکی استخوان در بیماران مبتلا به سندرم پوکی استخوان بسیار نادر و وراثتی استئوژنزیز ایمپرفکتا بررسی گردید و افزایش معنادار بیان ژن TNC در بیماران نسبت به افراد سالم مشاهده شد. این تغییر بیان میتواند نشاندهنده نقش احتمالی ژن TNC در روند استخوانزایی و پوکی استخوان طبیعی در دوران پیری باشد و این ژن را به عنوان یک بیومارکر تشخیصی معرفی کند.
سپاسگزاری
این مقاله برگرفته از پایاننامه دانشگاه اصفهان میباشد. نویسندگان تحقیق حاضر از دکتر محمد شبول به دلیل شناسایی بیماران و تهیه نمونههای انسانی از کشور اردن کمال تشکر را دارند.
حامی مالی: دانشگاه اصفهان
تعارض در منافع: وجود ندارد.
References:
1-Johnell O, Kanis JA. An Estimate of the Worldwide Prevalence and Disability Associated with Osteoporotic Fractures. Osteoporosis Int 2006; 17(12): 1726-33.
2-Kanis JA, Melton LJ, Christiansen C, Johnston CC, et al. The Diagnosis of Osteoporosis. The Lancet 2002; 359: 1929- 36.
3-Rodríguez-Rodero S, Fernández-Morera JL, Menéndez-Torre E, Calvanese V, Fernández AF, Fraga MF. Aging Genetics and Aging. Aging Dis 2011; 2(3): 186-95.
4-Huang S, Ng GC, You-Qiang S. Genetic Disorders Associated with Osteoporosis. Advances in Osteoporosis 2015: 19.
5-Chiquet-Ehrismann R, Chiquet M. Tenascins: Regulation and Putative Functions during Pathological Stress. J Pathol 2003; 200(4): 488-99.
6-Sato R, Fukuoka H, Yokohama-Tamaki T, Kaku M, Shibata SH. Immunohistochemical Localization of Tenascin-C in Rat Periodontal Ligament with Reference to Alveolar Bone Remodeling. Anat Sci Int 2016; 91(2): 196-206.
7-Li C, Li G, Liu M, Zhou T. Paracrine Effect of Inflammatory Cytokine-Activated Bone Marrow Mesenchymal Stem Cells and its Role in Osteoblast Function. J Biosci Bioeng 2016; 121(2): 213-9.
8-De Laporte L, Rice JJ, Tortelli F, Hubbell JA. Tenascin C Promiscuously Binds Growth Factors Via Its Fifth Fibronectin Type III-Like Domain. Plos One 2013; 8(4): E62076.
9-Wang A, Ding X, Sheng S, Yao Z. Bone Morphogenetic Protein Receptor in the Osteogenic Differentiation of Rat Bone Marrow Stromal Cells. Yonsei Med J 2010; 51(5): 740-5.
10-Shinohara Y, Okamoto K, Goh Y, Kiga N, Tojyo I, Fujita S. Inhibition of Fibrous Adhesion Formation in the Temporomandibular Joint of Tenascin-C Knockout Mice. Eur J Histochem 2014; 58(4): 263-70.
11-Sroga GE, Vashishth D. Effects of Bone Matrix Proteins on Fracture and Fragility in Osteoporosis. Curr Osteoporos Rep 2012; 10(2): 141-50.
12-Zhang D, Li X, Pi C, Cai L, Liu Y, Du W, et al. Osteoporosis-Decreased Extracellular Matrix Stiffness Impairs Connexin 43-Mediated Gap Junction Intercellular Communication in Osteocytes. Acta Biochim Biophys Sin (Shanghai) 2020; 52(5): 517-26.
13-Consensus A. Consensus Development Conference: Diagnosis, Prophylaxis, and Treatment of Osteoporosis. Am J Med 1993; 94(6): 646-50.
14-Demontiero O, Vidal C, Duque G. Aging and Bone Loss: New Insights for the Clinician. Ther Adv Musculoskel Dis 2012; 4(2): 61-76.
15-Hasegawa M, Hirata H, Sudo A, Kato K, Kawase D, Kinoshita N, et al. Tenascin-C Concentration in Synovial Fluid Correlates With Radiographic Progression of Knee Osteoarthritis. J Rheumatol 2004; 31(10): 2021-6.
16-Pazzaglia L, Conti A, Chiechi A, Novello C, Magagnoli G, Astolfi A, Pession A, et al. Differential Gene Expression in Classic Giant Cell Tumours of Bone: Tenascin C as Biological Risk Factor for Local Relapses and Metastases. Histopathology 2010; 57(1): 59-72.
17-Alford AI, Hankenson KD. Matricellular Proteins: Extracellular Modulators of Bone Development, Remodeling, And Regeneration. Bone 2006; 38(6): 749-57.
18-Chiquet-Ehrismann R. Tenascins. Int J Biochem Cell Biol 2004; 36: 986-90.
19-Järveläinen H, Sainio A, Koulu M, Wight TN, Penttinen R. Extracellular Matrix Molecules: Potential Targets in Pharmacotherapy. Pharmacological Reviews 2009; 61(2): 198-223.
20-Alford AI, Kozloff KM, Hankenson KD. Extracellular Matrix Networks in Bone Remodeling. Int J Biochem Cell Biol 2015; 65: 20-31.
21-Nyman JS, Makowski AJ. The Contribution of the Extracellular Matrix to the Fracture Resistance of Bone. Curr Osteoporos Rep 2012; 10(2): 169-77.
22-Mansouri R, Hay E, Marie PJ, Modrowski D. Role of Syndecan-2 in Osteoblast Biology and Pathology. Bonekey Reports 2015; 4.
23-Gao F, Xu F, Wu D, Cheng J, Xia P. Identification of Novel Genes Associated with Fracture Healing in Osteoporosis Induced by Krm2 Overexpression or Lrp5 Deficiency. Mol Med Rep 2017; 15(6): 3969-76.
24-De Sousa Brito FM, Butcher A, Pisconti A, Poulet B, et al. Syndecan-3 Enhances Anabolic Bone Formation through WNT Signalling. Biorxiv 2019: 846972.
25-Bertrand J, Stange R, Hidding H, Echtermeyer F, Nalesso G, Godmann L, et al. Syndecan 4 Supports Bone Fracture Repair, but Not Fetal Skeletal Development, In Mice. Arthritis & Rheumatism 2013; 65(3): 743-52.
26-Ozkan G, Ulusoy S, Guvercin B, Menteşe A, Karahan SC, Yavuz A. A New Player in Chronic Kidney Disease Mineral and Bone Disorder: Tenascin-C. Int J Artif Organs 2015; 38(9): 481-7.
27-Page TH, Charles PJ, Piccinini AM, Nicolaidou V, Taylor PC, Midwood KS. Raised Circulating Tenascin-C in Rheumatoid Arthritis. Arthritis Res Ther 2012; 14(6): R260.
28-Patel L, Sun W, Glasson SS, Morris EA, Flannery CR, Chockalingam PS. Tenascin-C Induces Inflammatory Mediators and Matrix Degradation in Osteoarthritic Cartilage. BMC Musculoskelet Disord 2011; 12: 164.
29-Chen Y, Chen ZF, He F. Tenascin-C Knockdown Suppresses Osteoblast Differentiation and Promotes Osteoporosis In Mice By Inhibiting Wnt Signaling. Nan Fang Yi Ke Da Xue Xue Bao J Southern Medical University 2016; 36(8): 1117-22.
1-Johnell O, Kanis JA. An Estimate of the Worldwide Prevalence and Disability Associated with Osteoporotic Fractures. Osteoporosis Int 2006; 17(12): 1726-33.
2-Kanis JA, Melton LJ, Christiansen C, Johnston CC, et al. The Diagnosis of Osteoporosis. The Lancet 2002; 359: 1929- 36.
3-Rodríguez-Rodero S, Fernández-Morera JL, Menéndez-Torre E, Calvanese V, Fernández AF, Fraga MF. Aging Genetics and Aging. Aging Dis 2011; 2(3): 186-95.
4-Huang S, Ng GC, You-Qiang S. Genetic Disorders Associated with Osteoporosis. Advances in Osteoporosis 2015: 19.
5-Chiquet-Ehrismann R, Chiquet M. Tenascins: Regulation and Putative Functions during Pathological Stress. J Pathol 2003; 200(4): 488-99.
6-Sato R, Fukuoka H, Yokohama-Tamaki T, Kaku M, Shibata SH. Immunohistochemical Localization of Tenascin-C in Rat Periodontal Ligament with Reference to Alveolar Bone Remodeling. Anat Sci Int 2016; 91(2): 196-206.
7-Li C, Li G, Liu M, Zhou T. Paracrine Effect of Inflammatory Cytokine-Activated Bone Marrow Mesenchymal Stem Cells and its Role in Osteoblast Function. J Biosci Bioeng 2016; 121(2): 213-9.
8-De Laporte L, Rice JJ, Tortelli F, Hubbell JA. Tenascin C Promiscuously Binds Growth Factors Via Its Fifth Fibronectin Type III-Like Domain. Plos One 2013; 8(4): E62076.
9-Wang A, Ding X, Sheng S, Yao Z. Bone Morphogenetic Protein Receptor in the Osteogenic Differentiation of Rat Bone Marrow Stromal Cells. Yonsei Med J 2010; 51(5): 740-5.
10-Shinohara Y, Okamoto K, Goh Y, Kiga N, Tojyo I, Fujita S. Inhibition of Fibrous Adhesion Formation in the Temporomandibular Joint of Tenascin-C Knockout Mice. Eur J Histochem 2014; 58(4): 263-70.
11-Sroga GE, Vashishth D. Effects of Bone Matrix Proteins on Fracture and Fragility in Osteoporosis. Curr Osteoporos Rep 2012; 10(2): 141-50.
12-Zhang D, Li X, Pi C, Cai L, Liu Y, Du W, et al. Osteoporosis-Decreased Extracellular Matrix Stiffness Impairs Connexin 43-Mediated Gap Junction Intercellular Communication in Osteocytes. Acta Biochim Biophys Sin (Shanghai) 2020; 52(5): 517-26.
13-Consensus A. Consensus Development Conference: Diagnosis, Prophylaxis, and Treatment of Osteoporosis. Am J Med 1993; 94(6): 646-50.
14-Demontiero O, Vidal C, Duque G. Aging and Bone Loss: New Insights for the Clinician. Ther Adv Musculoskel Dis 2012; 4(2): 61-76.
15-Hasegawa M, Hirata H, Sudo A, Kato K, Kawase D, Kinoshita N, et al. Tenascin-C Concentration in Synovial Fluid Correlates With Radiographic Progression of Knee Osteoarthritis. J Rheumatol 2004; 31(10): 2021-6.
16-Pazzaglia L, Conti A, Chiechi A, Novello C, Magagnoli G, Astolfi A, Pession A, et al. Differential Gene Expression in Classic Giant Cell Tumours of Bone: Tenascin C as Biological Risk Factor for Local Relapses and Metastases. Histopathology 2010; 57(1): 59-72.
17-Alford AI, Hankenson KD. Matricellular Proteins: Extracellular Modulators of Bone Development, Remodeling, And Regeneration. Bone 2006; 38(6): 749-57.
18-Chiquet-Ehrismann R. Tenascins. Int J Biochem Cell Biol 2004; 36: 986-90.
19-Järveläinen H, Sainio A, Koulu M, Wight TN, Penttinen R. Extracellular Matrix Molecules: Potential Targets in Pharmacotherapy. Pharmacological Reviews 2009; 61(2): 198-223.
20-Alford AI, Kozloff KM, Hankenson KD. Extracellular Matrix Networks in Bone Remodeling. Int J Biochem Cell Biol 2015; 65: 20-31.
21-Nyman JS, Makowski AJ. The Contribution of the Extracellular Matrix to the Fracture Resistance of Bone. Curr Osteoporos Rep 2012; 10(2): 169-77.
22-Mansouri R, Hay E, Marie PJ, Modrowski D. Role of Syndecan-2 in Osteoblast Biology and Pathology. Bonekey Reports 2015; 4.
23-Gao F, Xu F, Wu D, Cheng J, Xia P. Identification of Novel Genes Associated with Fracture Healing in Osteoporosis Induced by Krm2 Overexpression or Lrp5 Deficiency. Mol Med Rep 2017; 15(6): 3969-76.
24-De Sousa Brito FM, Butcher A, Pisconti A, Poulet B, et al. Syndecan-3 Enhances Anabolic Bone Formation through WNT Signalling. Biorxiv 2019: 846972.
25-Bertrand J, Stange R, Hidding H, Echtermeyer F, Nalesso G, Godmann L, et al. Syndecan 4 Supports Bone Fracture Repair, but Not Fetal Skeletal Development, In Mice. Arthritis & Rheumatism 2013; 65(3): 743-52.
26-Ozkan G, Ulusoy S, Guvercin B, Menteşe A, Karahan SC, Yavuz A. A New Player in Chronic Kidney Disease Mineral and Bone Disorder: Tenascin-C. Int J Artif Organs 2015; 38(9): 481-7.
27-Page TH, Charles PJ, Piccinini AM, Nicolaidou V, Taylor PC, Midwood KS. Raised Circulating Tenascin-C in Rheumatoid Arthritis. Arthritis Res Ther 2012; 14(6): R260.
28-Patel L, Sun W, Glasson SS, Morris EA, Flannery CR, Chockalingam PS. Tenascin-C Induces Inflammatory Mediators and Matrix Degradation in Osteoarthritic Cartilage. BMC Musculoskelet Disord 2011; 12: 164.
29-Chen Y, Chen ZF, He F. Tenascin-C Knockdown Suppresses Osteoblast Differentiation and Promotes Osteoporosis In Mice By Inhibiting Wnt Signaling. Nan Fang Yi Ke Da Xue Xue Bao J Southern Medical University 2016; 36(8): 1117-22.
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |







